写在前面
学习一个软件最好的办法便是啃它的官方文档。本着自己学习、共享别人的情绪,共享官方文档的中文教程。软件或许随时更新,建议配合官方文档一起阅读。引荐先按次序阅读往期内容:
文献篇:
1.文献阅读:运用 CellChat 推理和剖析细胞-细胞通讯
::: block-1
目录
- 0 加载所需的库
- 1 Part I: CellChat 方针的数据输入、处理、和初始化
- 2 Part II: 揣度细胞间通讯网络
- 3 Part III: 可视化细胞间通讯网络
- 4 Part IV: 细胞间通讯网络系统剖析
- 5 Part V: 保存 CellChat 方针
- 6 Part VI: 经过交互式 CellChat Explorer 探究细胞间通讯 :::
官网教程:
https://htmlpreview.github.io/?https://github.com/jinworks/CellChat/blob/master/tutorial/CellChat-vignette.html
这个教程概述了运用 CellChat 对单个数据集进行细胞间通讯网络的推理、剖析和可视化的步骤。咱们经过将 CellChat 应用于来自患者的病变(LS,患病)人类皮肤细胞的 scRNA-seq 数据来展现 CellChat 的多种功用。
CellChat 需求细胞的基因表达数据作为用户输入,并经过将基因表达与信号配体(ligands)、受体(receptors)及其辅助因子(cofactors)之间彼此效果的先验知识相结合来模仿细胞间通讯的概率。
在揣度细胞间通讯网络后,CellChat 供给进一步数据探究、剖析和可视化的功用。
0 加载所需的库
library(CellChat)
library(patchwork)
options(stringsAsFactors = FALSE)
# reticulate::use_python("/Users/suoqinjin/anaconda3/bin/python", required=T)
1 Part I: CellChat 方针的数据输入、处理、和初始化
CellChat 需求两个用户输入:一个是细胞的基因表达数据,另一个是用户分配的细胞标签。
1.1 准备 CellChat 剖析所需的输入数据
关于基因表达数据矩阵,行名为 genes,列名为 cells。Normalized data (e.g., library-size normalization and then log-transformed with a pseudocount of 1) 作为 CellChat 剖析的输入。假如用户供给 count data,咱们供给一个 normalizeData 函数来核算 library size,然后进行 log-transformed。
关于 cell group 信息,需求一个带有行名的 dataframe 作为 CellChat 的输入。
除了承受 count data 矩阵作为输入外,咱们还供给了怎么从其他现有的单细胞剖析工具包(包括 Seurat、SingleCellExperiment 和 Scanpy)准备 CellChat 输入文件的说明。当 normalized count data 和 meta data 可用时,请按照选项 A 开始准备输入数据;当有 Seurat 方针可用时,请按照选项 B 开始;当有 SingleCellExperiment 方针可用时,请按照选项 C 开始;当有 Anndata 方针可用时,请按照选项 D 开始。详细信息请参阅 tutorial on Interface_with_other_single-cell_analysis_toolkits。
这儿运用选项 A 导入数据
(A) Starting from a count data matrix
ptm = Sys.time()
# 这儿咱们加载了一个 scRNA-seq 数据矩阵以及其相关的 cell meta data
# 这是来自两种生物条件的合并数据:normal 和 diseases
# 数据下载链接:https://figshare.com/articles/dataset/scRNA-seq_data_of_human_skin_from_patients_with_atopic_dermatitis/24470719
load("data_humanSkin_CellChat.rda")
data.input = data_humanSkin$data # normalized data matrix
meta = data_humanSkin$meta # a dataframe with rownames containing cell mata data
cell.use = rownames(meta)[meta$condition == "LS"] # extract the cell names from disease data
# Subset the input data for CelChat analysis
data.input = data.input[, cell.use]
meta = meta[cell.use, ]
unique(meta$labels) # check the cell labels
1.2 创立一个 CellChat 方针
用户能够从一个 data matrix、Seurat、SingleCellExperiment、AnnData 方针创立一个新的 CellChat 方针。假如输入是 Seurat 或 SingleCellExperiment 方针,则默许运用方针中的 metadata,用户有必要供给 group.by 来界说 cell groups。例如,关于 Seurat 方针中的默许 cell identities,group.by = "ident"。
按照以下选项之一创立一个 CellChat 方针:选项 A:运用数字基因表达矩阵和细胞标签信息作为输入,选项 B:运用 Seurat 方针作为输入,选项 C:运用 SingleCellExperiment 方针作为输入,选项 D:运用 AnnData 方针作为输入
留意:假如用户加载了从前核算的 CellChat 方针(版别 < 0.5.0),请经过 updateCellChat 更新方针。
这儿运用选项 A 创立一个 CellChat 方针
(A) Starting from the digital gene expression matrix and cell label information
cellchat <- createCellChat(object = data.input, meta = meta, group.by = "labels")
假如在创立 CellChat 方针时未增加 cell meta 数据信息,用户也能够在之后运用 addMeta 增加它,并运用 setIdent 设置默许细胞身份。
cellchat <- addMeta(cellchat, meta = meta)
cellchat <- setIdent(cellchat, ident.use = "labels") # set "labels" as default cell identity
levels(cellchat@idents) # show factor levels of the cell labels
groupSize <- as.numeric(table(cellchat@idents)) # number of cells in each cell group
1.3 设置配体-受体彼此效果数据库
在用户能够运用 CellChat 揣度细胞间通讯之前,他们需求设置配体-受体(ligand-receptor)彼此效果数据库,并确认过表达的配体(ligands)或受体(receptors)。
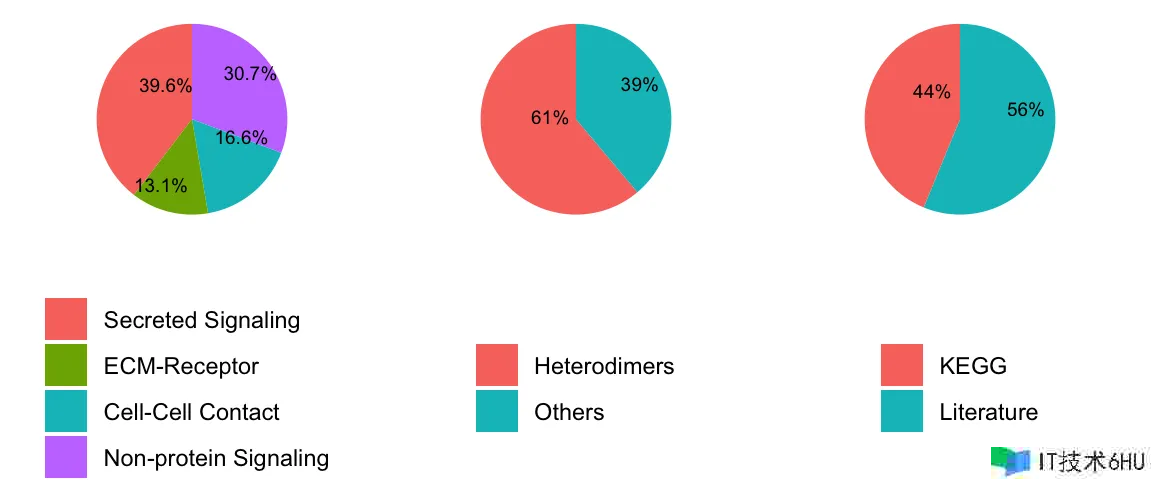
CellChatDB 数据库是一个人工管理的数据库,包括文献支撑的人类和小鼠的配体-受体彼此效果。CellChatDB v2 包括约 3,300 个经过验证的分子彼此效果,包括约 40% 的自排泄/旁排泄信号彼此效果,约 17% 的细胞外基质(ECM)-受体彼此效果,约 13% 的细胞-细胞触摸彼此效果和约 30% 的非蛋白信号传导。与 CellChatDB v1 比较,CellChatDB v2 增加了 1000 多个蛋白质和非蛋白质彼此效果,例如代谢和突触信号传导。值得留意的是,关于与 scRNA-seq 中丈量的基因直接相关的分子,CellChat v2 经过这些分子的要害介质或酶来估算配体和受体的表达,以便经过非蛋白质介导的潜在通讯。
CellChatDB v2 还增加了配体-受体对的其他功用注释,例如 UniProtKB 要害词(包括生物进程、分子功用、功用类别、疾病等)、亚细胞方位和与神经递质的相关性。
用户能够经过增加自己筛选过的配体-受体对来更新 CellChatDB。请查看tutorial on updating the ligand-receptor interaction database CellChatDB。
在剖析人类样本时,请运用数据库 CellChatDB.human;在剖析小鼠样本时,请运用数据库 CellChatDB.mouse。CellChatDB 将配体-受体对分类为不同类型,包括“排泄信号(Secreted Signaling)”、“细胞外基质-受体(ECM-Receptor)”、“细胞-细胞触摸(Cell-Cell Contact)”和“非蛋白质信号(Non-protein Signaling)”。默许情况下,“非蛋白质信号(Non-protein Signaling)”不被运用。
CellChatDB <- CellChatDB.human # use CellChatDB.mouse if running on mouse data
showDatabaseCategory(CellChatDB)
# 显现数据库结构
dplyr::glimpse(CellChatDB$interaction)
#> Rows: 3,234
#> Columns: 28
#> $ interaction_name <chr> "TGFB1_TGFBR1_TGFBR2", "TGFB2_TGFBR1_TGFBR2",…
#> $ pathway_name <chr> "TGFb", "TGFb", "TGFb", "TGFb", "TGFb", "TGFb…
#> $ ligand <chr> "TGFB1", "TGFB2", "TGFB3", "TGFB1", "TGFB1", …
#> $ receptor <chr> "TGFbR1_R2", "TGFbR1_R2", "TGFbR1_R2", "ACVR1…
#> $ agonist <chr> "TGFb agonist", "TGFb agonist", "TGFb agonist…
#> $ antagonist <chr> "TGFb antagonist", "TGFb antagonist", "TGFb a…
#> $ co_A_receptor <chr> "", "", "", "", "", "", "", "", "", "", "", "…
#> $ co_I_receptor <chr> "TGFb inhibition receptor", "TGFb inhibition …
#> $ evidence <chr> "KEGG: hsa04350", "KEGG: hsa04350", "KEGG: hs…
#> $ annotation <chr> "Secreted Signaling", "Secreted Signaling", "…
#> $ interaction_name_2 <chr> "TGFB1 - (TGFBR1 TGFBR2)", "TGFB2 - (TGFBR1 T…
#> $ is_neurotransmitter <lgl> FALSE, FALSE, FALSE, FALSE, FALSE, FALSE, FAL…
#> $ ligand.symbol <chr> "TGFB1", "TGFB2", "TGFB3", "TGFB1", "TGFB1", …
#> $ ligand.family <chr> "TGF-beta", "TGF-beta", "TGF-beta", "TGF-beta…
#> $ ligand.location <chr> "Extracellular matrix, Secreted, Extracellula…
#> $ ligand.keyword <chr> "Disease variant, Signal, Reference proteome,…
#> $ ligand.secreted_type <chr> "growth factor", "growth factor", "cytokine;g…
#> $ ligand.transmembrane <lgl> FALSE, FALSE, TRUE, FALSE, FALSE, FALSE, FALS…
#> $ receptor.symbol <chr> "TGFBR2, TGFBR1", "TGFBR2, TGFBR1", "TGFBR2, …
#> $ receptor.family <chr> "Protein kinase superfamily, TKL Ser/Thr prot…
#> $ receptor.location <chr> "Cell membrane, Secreted, Membrane raft, Cell…
#> $ receptor.keyword <chr> "Membrane, Secreted, Disulfide bond, Kinase, …
#> $ receptor.surfaceome_main <chr> "Receptors", "Receptors", "Receptors", "Recep…
#> $ receptor.surfaceome_sub <chr> "Act.TGFB;Kinase", "Act.TGFB;Kinase", "Act.TG…
#> $ receptor.adhesome <chr> "", "", "", "", "", "", "", "", "", "", "", "…
#> $ receptor.secreted_type <chr> "", "", "", "", "", "", "", "", "", "", "", "…
#> $ receptor.transmembrane <lgl> TRUE, TRUE, TRUE, TRUE, TRUE, TRUE, TRUE, TRU…
#> $ version <chr> "CellChatDB v1", "CellChatDB v1", "CellChatDB…
# 运用 CellChatDB 的子集进行细胞间通讯剖析
CellChatDB.use <- subsetDB(CellChatDB, search = "Secreted Signaling", key = "annotation") # use Secreted Signaling
# Only uses the Secreted Signaling from CellChatDB v1
# CellChatDB.use <- subsetDB(CellChatDB, search = list(c("Secreted Signaling"), c("CellChatDB v1")), key = c("annotation", "version"))
# use all CellChatDB except for "Non-protein Signaling" for cell-cell communication analysis
# CellChatDB.use <- subsetDB(CellChatDB)
# use all CellChatDB for cell-cell communication analysis
# CellChatDB.use <- CellChatDB # simply use the default CellChatDB. We do not suggest to use it in this way because CellChatDB v2 includes "Non-protein Signaling" (i.e., metabolic and synaptic signaling).
# 在方针中设置运用的数据库
cellchat@DB <- CellChatDB.use
1.4 预处理表达数据以进行细胞间通讯剖析
为了揣度特定于细胞状况的通讯,CellChat 在一个 cell group 中辨认出过表达的配体(ligands)或受体(receptors),然后在配体或受体过表达时辨认过表达的配体-受体(ligand-receptor)彼此效果。
CellChat 还供给了将基因表达数据投射到 protein-protein interaction(PPI) 网络上的功用。详细来说,a diffusion process 用于依据在高置信度试验验证的 PPI 网络中界说的街坊基因的表达值来平滑基因的表达值。此功用在剖析具有浅测序深度的单细胞数据时十分有用,由于投射减少了信号基因的丢掉效应,特别是关于配体/受体亚基的零表达。人们或许会忧虑这种 diffusion process 或许引进 artifact,可是,它只会引进十分微弱的通讯。默许情况下 CellChat 运用 raw data(i.e., object@data.signaling)而不是 projected data。为了运用 projected data,用户需求在运转 computeCommunProb() 之前运转 projectData,并在运转 computeCommunProb 时设置 raw.use = TRUE。
# 对信号基因的表达数据取子集以节省核算成本
cellchat <- subsetData(cellchat) # This step is necessary even if using the whole database
future::plan("multisession", workers = 4) # do parallel
cellchat <- identifyOverExpressedGenes(cellchat)
cellchat <- identifyOverExpressedInteractions(cellchat)
execution.time = Sys.time() - ptm
print(as.numeric(execution.time, units = "secs"))
# 将基因表达数据投射到 PPI 上(可选:运转时,用户应在函数 computeCommunProb() 中设置 raw.use = FALSE 以运用投射数据)
# cellchat <- projectData(cellchat, PPI.human)
2 Part II: 揣度细胞间通讯网络
CellChat 经过为每个交互分配一个概率值并履行排列测验来揣度具有生物学含义的细胞间通讯。CellChat 经过运用质量效果定律将基因表达与信号配体、受体及其辅因子之间彼此效果的从前已知知识相结合来模仿细胞间通讯的或许性。
留意:揣度的配体-受体对数目明显取决于核算每个 cell group 的平均基因表达量的办法。默许情况下,CellChat 运用一种称为 “trimean” 的核算上稳健的均值办法,与其他办法比较,它发生的交互更少。然而,咱们发现 CellChat 在猜测更强的彼此效果方面体现良好,这关于缩小彼此效果范围以进行进一步的试验验证十分有帮助。在 computeCommunProb 中,咱们供给了运用其他办法,例如 5% 和 10% truncated mean 来核算平均基因表达的选项。值得留意的是,“trimean”近似于 25% truncated mean,这意味着假如一组中表达细胞的百分比小于 25%,则平均基因表达为零。要运用 10% truncated mean,用户能够设置 type = "truncatedMean" 和 trim = 0.1。为了确认 trim 的合适值,CellChat 供给了一个函数 computeAveExpr 能够帮助查看感兴趣的信号基因的平均表达,例如 computeAveExpr(cellchat, features = c("CXCL12","CXCR4"), type = "truncatedMean", trim = 0.1)。因此,假如在研讨的生物进程中未猜测到已知的信号通路,用户能够尝试运用较低的 trim 值来改动核算每个 cell group 平均基因表达量的办法。
在剖析未分类的单细胞转录组时,假设丰厚的细胞群往往比稀有细胞群发送更强的信号,CellChat 还能够在概率核算中考虑每个细胞群中细胞份额的影响。用户能够设置 population.size = TRUE。
2.1 核算通讯概率并揣度细胞通讯网络
ptm = Sys.time()
cellchat <- computeCommunProb(cellchat, type = "triMean")
这项剖析的要害参数是 type,用于核算每个细胞组平均基因表达量的办法。默许情况下,type = "triMean",会发生较少但较强的彼此效果。当设置 type = "truncatedMean" 时,应为 trim 分配一个值,这会发生更多的彼此效果。请在上面详细查看核算每个细胞组平均基因表达量的办法。
用户能够在某些细胞组中仅有少数细胞时过滤掉细胞间通讯。默许情况下,每个细胞组中用于细胞间通讯的最小细胞数为 10。
cellchat <- filterCommunication(cellchat, min.cells = 10)
2.2 将揣度的细胞通讯网络提取为一个数据框
CellChat 供给了一个函数 subsetCommunication 来轻松拜访感兴趣的揣度细胞间通讯。例如,
-
df.net <- subsetCommunication(cellchat)回来一个数据框,其间包括配体/受体等级的一切揣度的细胞间通讯。设置slot.name = "netP"以拜访信号通路等级的揣度通讯 -
df.net <- subsetCommunication(cellchat, sources.use = c(1,2), targets.use = c(4,5))给出了从 cell groups 1 和 2 到 cell groups 4 和 5。
-
df.net <- subsetCommunication(cellchat, signaling = c("WNT", "TGFb"))给出了由信号 WNT 和 TGFb 介导的揣度细胞间通讯。
2.3 在信号通路水平上揣度细胞间通讯
CellChat 经过汇总与每个信号通路相关的一切配体-受体彼此效果的通讯概率,核算信号通路水平上的通讯概率。
留意:每个配体-受体对和每个信号通路的揣度细胞间通讯网络别离存储在 slot `net` 和 `netP` 中。
cellchat <- computeCommunProbPathway(cellchat)
2.4 核算聚合的细胞间通讯网络
咱们能够经过核算连接数或汇总通讯概率来核算聚合的细胞间通讯网络。用户还能够经过设置 sources.use 和 targets.use 来核算 cell groups 子集之间的聚合网络。
cellchat <- aggregateNet(cellchat)
execution.time = Sys.time() - ptm
print(as.numeric(execution.time, units = "secs"))
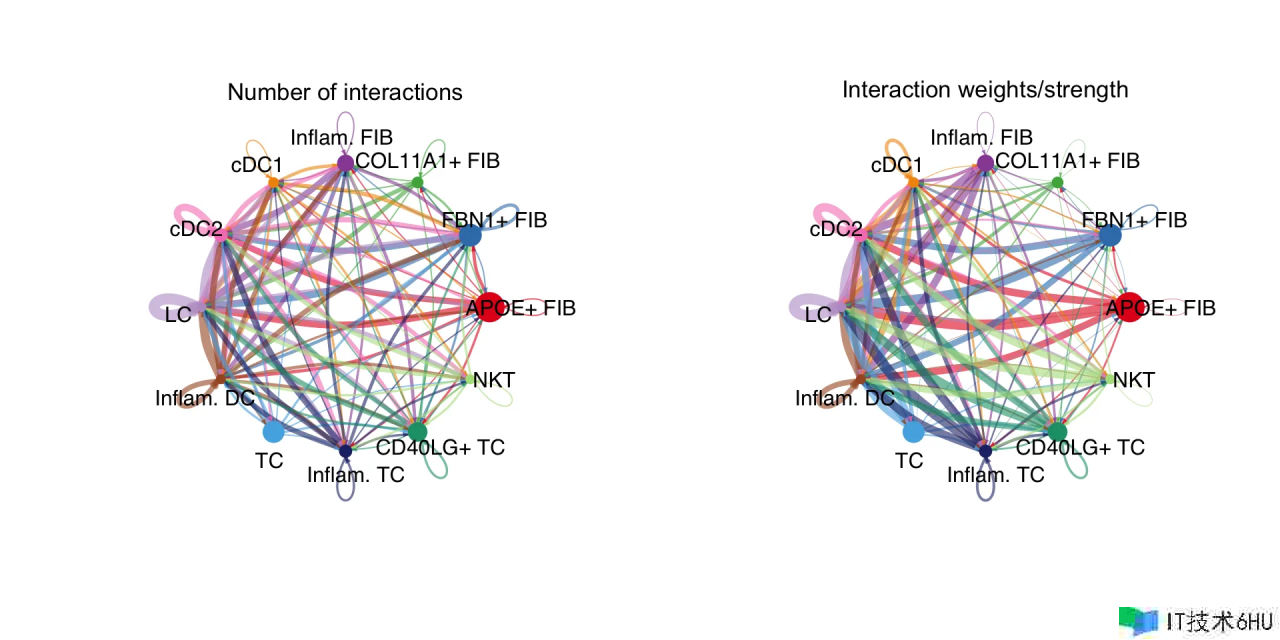
CellChat 还能够可视化聚合的细胞间通讯网络。例如,运用 circle plot 显现任意两个 cell groups 之间的交互次数或总交互强度(权重)。
ptm = Sys.time()
groupSize <- as.numeric(table(cellchat@idents))
par(mfrow = c(1,2), xpd=TRUE)
netVisual_circle(cellchat@net$count, vertex.weight = groupSize, weight.scale = T, label.edge= F, title.name = "Number of interactions")
netVisual_circle(cellchat@net$weight, vertex.weight = groupSize, weight.scale = T, label.edge= F, title.name = "Interaction weights/strength")
由于杂乱的细胞-细胞通讯网络,咱们能够查看从每个 cell group 发送的信号。在这儿咱们还控制参数 edge.weight.max 以便咱们能够比较不同网络之间的边权重 (edge weights)。
mat <- cellchat@net$weight
par(mfrow = c(3,4), xpd=TRUE)
for (i in 1:nrow(mat)) {
mat2 <- matrix(0, nrow = nrow(mat), ncol = ncol(mat), dimnames = dimnames(mat))
mat2[i, ] <- mat[i, ]
netVisual_circle(mat2, vertex.weight = groupSize, weight.scale = T, edge.weight.max = max(mat), label.edge= F, title.name = rownames(mat)[i])
}
3 Part III: 可视化细胞间通讯网络
在揣度细胞间通讯网络后,CellChat 为进一步的数据探究、剖析和可视化供给了各种功用。详细有:
- 它供给了多种可视化细胞间通讯网络的办法,包括层次图、圆图、弦图和气泡图。
- 它供给了一种易于运用的工具,用于提取和可视化揣度网络的高阶信息。例如,它能够猜测细胞群的首要信号输入和输出,以及这些细胞群和信号怎么和谐在一起发挥功用。
- 它能够经过结合交际网络剖析、形式辨认和流形学习办法,运用集成办法定量表征和比较揣度的细胞间通讯网络。
3.1 运用层次图、圆图或弦图可视化每个信号通路
Hierarchy plot: 用户应该界说 vertex.receiver,它是一个 numeric vector,给出了 cell groups 的索引作为层次结构图左边的方针。该层次图由两个部分组成:左边部分显现了对某些感兴趣的细胞群(即界说的 vertex.receiver)的自排泄和旁排泄信号,右侧部分显现了对数据会集其余细胞群的自排泄和旁排泄信号。因此,层次结构图供给了一种信息丰厚且直观的办法来可视化感兴趣的细胞群之间的自排泄和旁排泄信号通讯。例如,在研讨成纤维细胞和免疫细胞之间的细胞间通讯时,用户能够将 vertex.receiver 界说为一切成纤维细胞群。
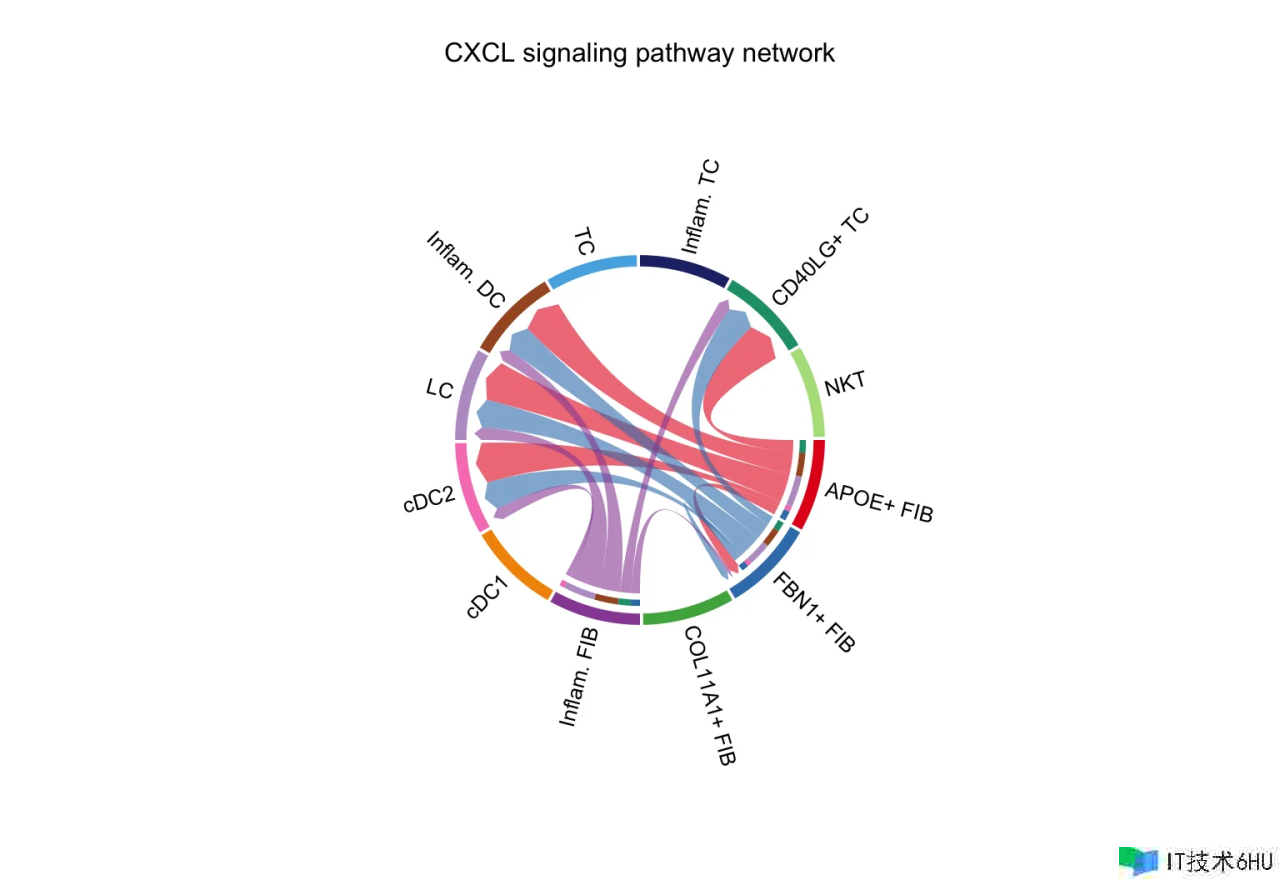
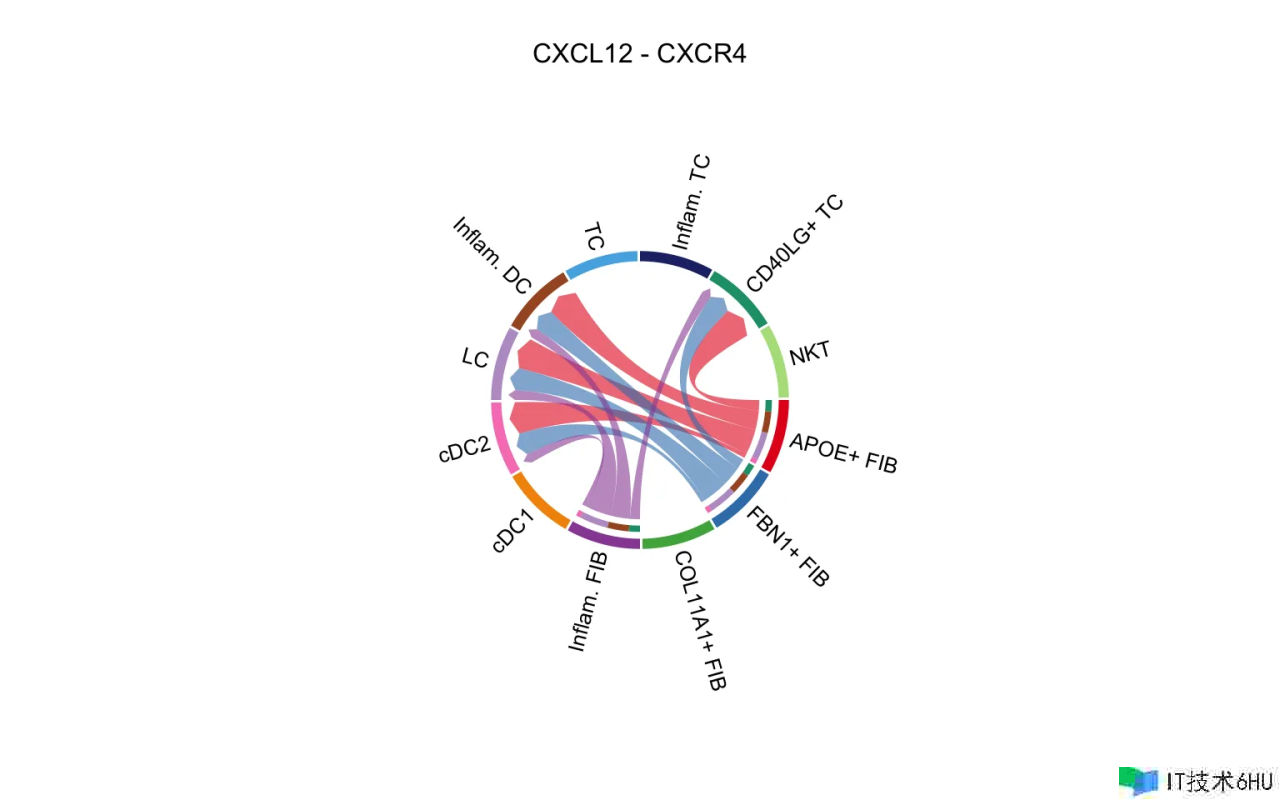
Chord diagram: CellChat 供给了两个函数 netVisual_chord_cell 和 netVisual_chord_gene 用于可视化不同意图和不同层次的细胞间通讯。netVisual_chord_cell 用于可视化不同细胞群之间的细胞间通讯(弦图中每个扇区为一个细胞群),netVisual_chord_gene 用于可视化由多个配体-受体或信号通路介导的细胞间通讯(其间弦图中的每个部分都是配体、受体或信号通路)。
Explnations of edge color/weight, node color/size/shape: 在一切可视化图中,edge colors 与作为发送者的 sources 一致,edge weights 与交互强度成正比。edge 线越粗标明信号越强。 在 Hierarchy plot 和 Circle plot 中,圆形巨细与每个 cell group 中的细胞数成正比。 在 hierarchy plot 中,实心圆和空心圆别离代表 source 和 target。在 Chord diagram 中,内部较细的条形颜色标明从相应外部条形接纳信号的方针。内部条形巨细与方针接纳到的信号强度成正比。这样的内部末节有助于解说杂乱的和弦图。请留意,关于某些 cell group,存在一些没有任何和弦的内部末节,请疏忽它,由于这是 circlize 包没有解决的问题。
Visualization of cell-cell communication at different levels: 能够运用 netVisual_aggregate 可视化揣度的信号通路通讯网络,并运用 netVisual_individual 可视化与该信号通路相关的各个 L-R 对的揣度通讯网络。
这儿咱们以一个信号通路的输入为例。一切显现重要通讯的信号通路都能够经过 cellchat@netP$pathways 拜访。
pathways.show <- c("CXCL")
# Hierarchy plot
# Here we define `vertex.receive` so that the left portion of the hierarchy plot shows signaling to fibroblast and the right portion shows signaling to immune cells
vertex.receiver = seq(1,4) # a numeric vector.
netVisual_aggregate(cellchat, signaling = pathways.show, vertex.receiver = vertex.receiver)
# Circle plot
par(mfrow=c(1,1))
netVisual_aggregate(cellchat, signaling = pathways.show, layout = "circle")
# Chord diagram
par(mfrow=c(1,1))
netVisual_aggregate(cellchat, signaling = pathways.show, layout = "chord")
# Heatmap
par(mfrow=c(1,1))
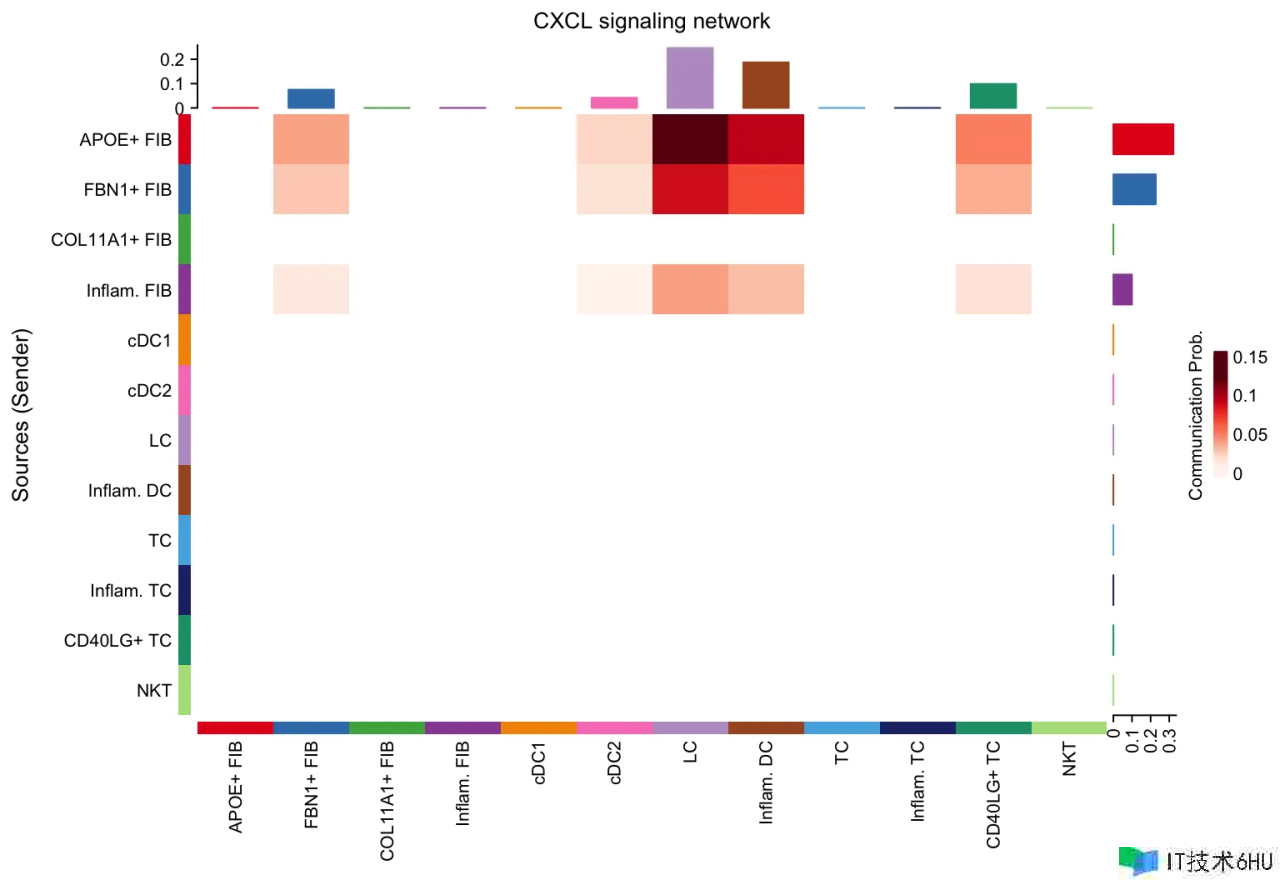
netVisual_heatmap(cellchat, signaling = pathways.show, color.heatmap = "Reds")
#> Do heatmap based on a single object
关于 chord diagram,CellChat 有一个独立的函数 netVisual_chord_cell,能够经过 circlize 包中调整不同的参数来灵活可视化通讯网络。例如,咱们能够界说一个命名的特征向量 group 来创立多组和弦图,例如,将细胞簇分组为不同的细胞类型。
# Chord diagram
group.cellType <- c(rep("FIB", 4), rep("DC", 4), rep("TC", 4)) # grouping cell clusters into fibroblast, DC and TC cells
names(group.cellType) <- levels(cellchat@idents)
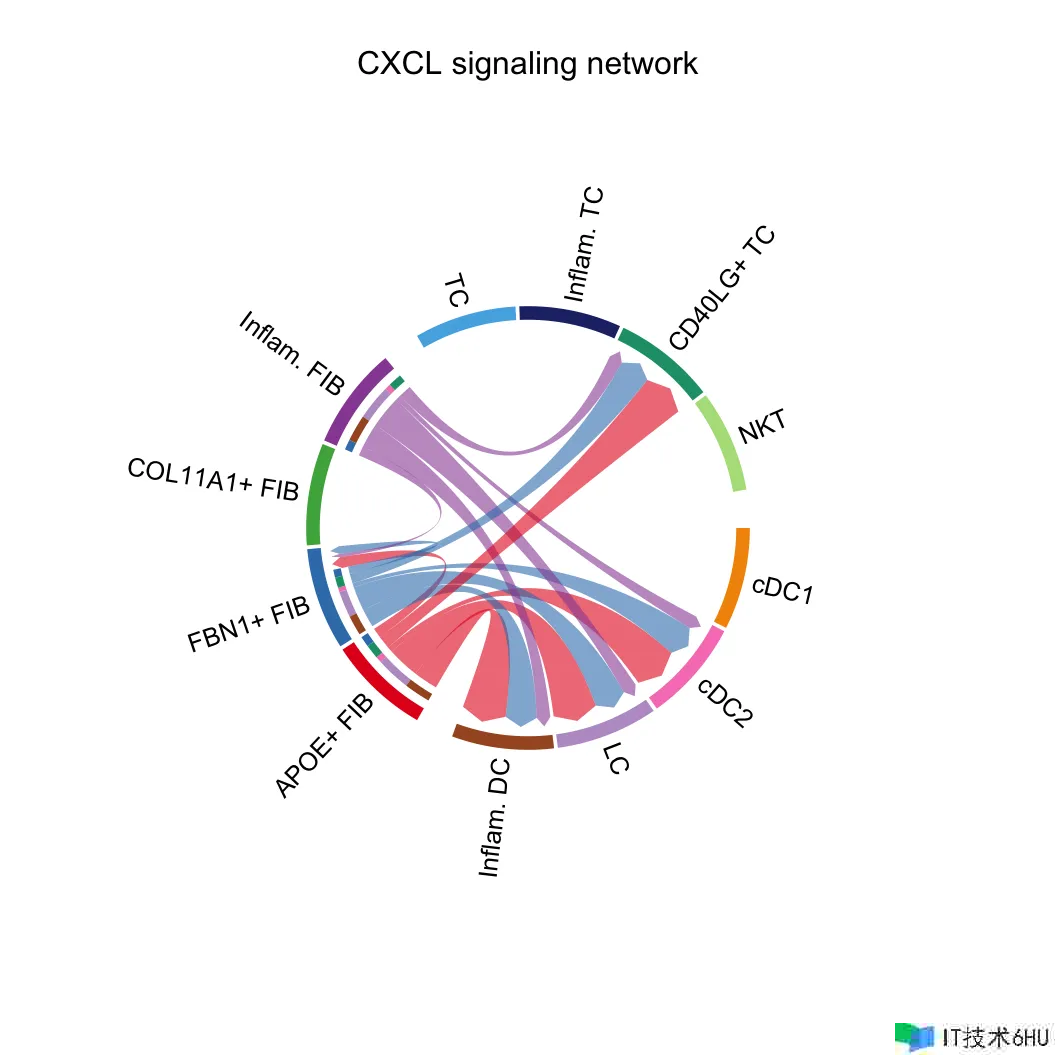
netVisual_chord_cell(cellchat, signaling = pathways.show, group = group.cellType, title.name = paste0(pathways.show, " signaling network"))
#> Plot the aggregated cell-cell communication network at the signaling pathway level
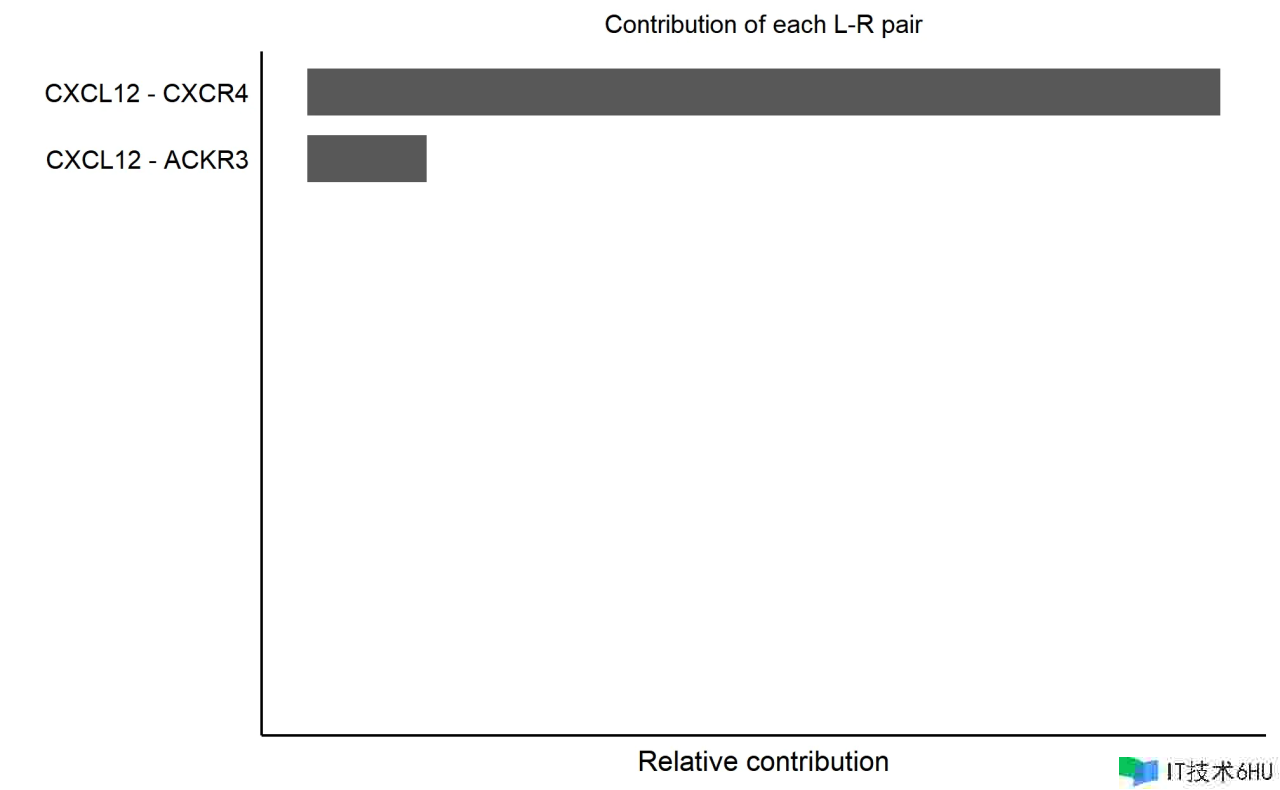
核算每个配体-受体对对整个信号通路的奉献,并可视化由单个配体-受体对介导的细胞间通讯
netAnalysis_contribution(cellchat, signaling = pathways.show)
咱们还能够可视化由单个配体-受体对介导的细胞间通讯。咱们供给了一个函数 extractEnrichedLR 来提取给定信号通路的一切重要彼此效果(L-R pairs)和相关信号基因。
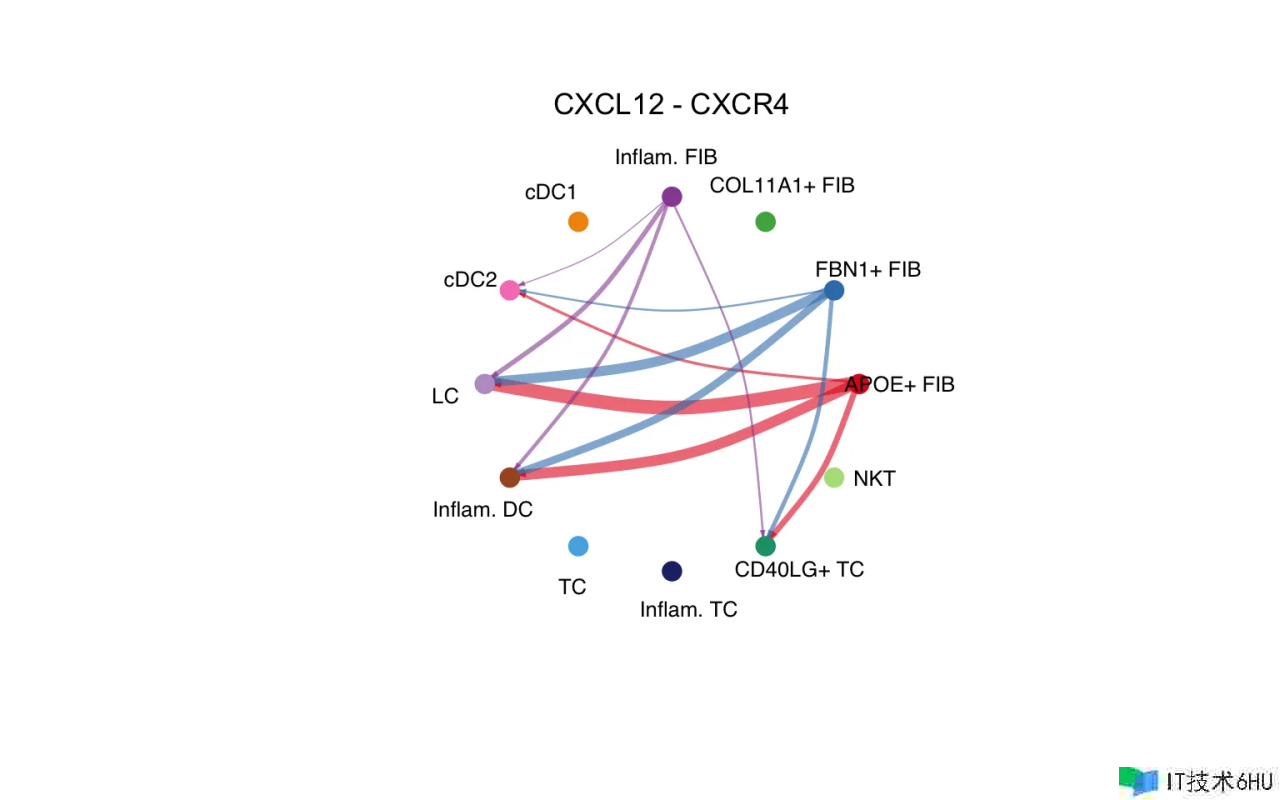
pairLR.CXCL <- extractEnrichedLR(cellchat, signaling = pathways.show, geneLR.return = FALSE)
LR.show <- pairLR.CXCL[1,] # show one ligand-receptor pair
# Hierarchy plot
vertex.receiver = seq(1,4) # a numeric vector
netVisual_individual(cellchat, signaling = pathways.show, pairLR.use = LR.show, vertex.receiver = vertex.receiver)
#> [[1]]
# Circle plot
netVisual_individual(cellchat, signaling = pathways.show, pairLR.use = LR.show, layout = "circle")
# Chord diagram
netVisual_individual(cellchat, signaling = pathways.show, pairLR.use = LR.show, layout = "chord")
主动保存一切揣度网络的图以便快速探究
在实际运用中,用户能够运用’for…loop’来主动保存一切揣度的网络,以便运用netVisual进行快速探究。netVisual支撑svg、png和pdf格局的输出。
# Access all the signaling pathways showing significant communications
pathways.show.all <- cellchat@netP$pathways
# check the order of cell identity to set suitable vertex.receiver
levels(cellchat@idents)
vertex.receiver = seq(1,4)
for (i in 1:length(pathways.show.all)) {
# Visualize communication network associated with both signaling pathway and individual L-R pairs
netVisual(cellchat, signaling = pathways.show.all[i], vertex.receiver = vertex.receiver, layout = "hierarchy")
# Compute and visualize the contribution of each ligand-receptor pair to the overall signaling pathway
gg <- netAnalysis_contribution(cellchat, signaling = pathways.show.all[i])
ggsave(filename=paste0(pathways.show.all[i], "_L-R_contribution.pdf"), plot=gg, width = 3, height = 2, units = 'in', dpi = 300)
}
3.2 可视化由多个配体-受体或信号通路介导的细胞间通讯
CellChat 还能够运用函数 netVisual_bubble(选项 A)和 netVisual_chord_gene(选项 B)显现由配体-受体对和信号通路介导的一切显著彼此效果,以及用户从某些细胞组到其他细胞组供给的彼此效果。
(A) Bubble plot
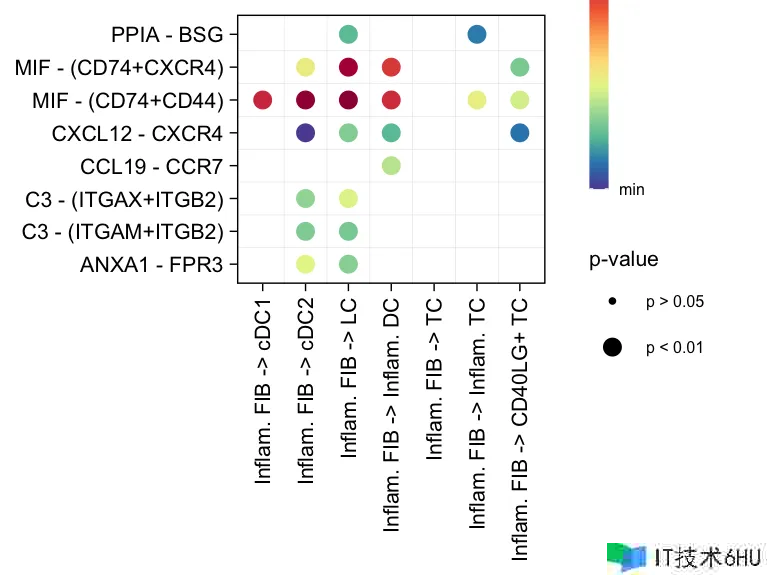
咱们还能够运用 netVisual_bubble 显现从一些 cell groups 到其他 cell groups 的一切重要彼此效果(L-R pairs)。
# (1) show all the significant interactions (L-R pairs) from some cell groups (defined by 'sources.use') to other cell groups (defined by 'targets.use')
netVisual_bubble(cellchat, sources.use = 4, targets.use = c(5:11), remove.isolate = FALSE)
#> Comparing communications on a single object
# (2) show all the significant interactions (L-R pairs) associated with certain signaling pathways
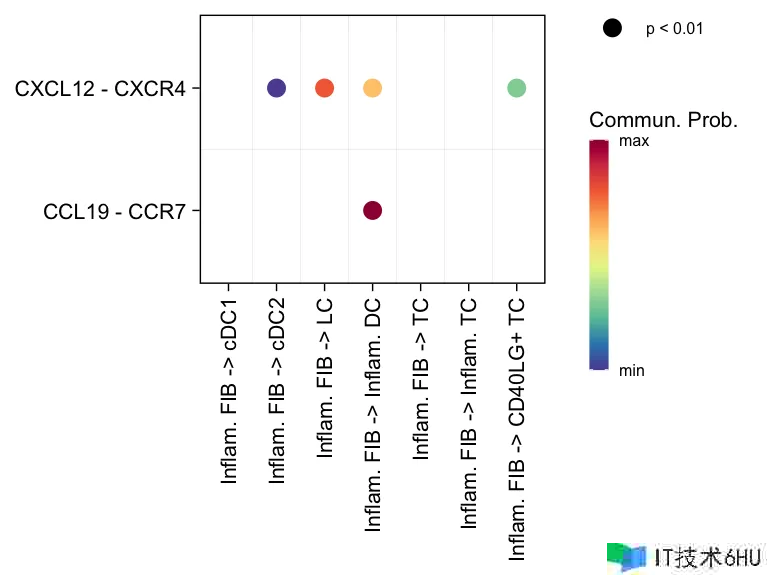
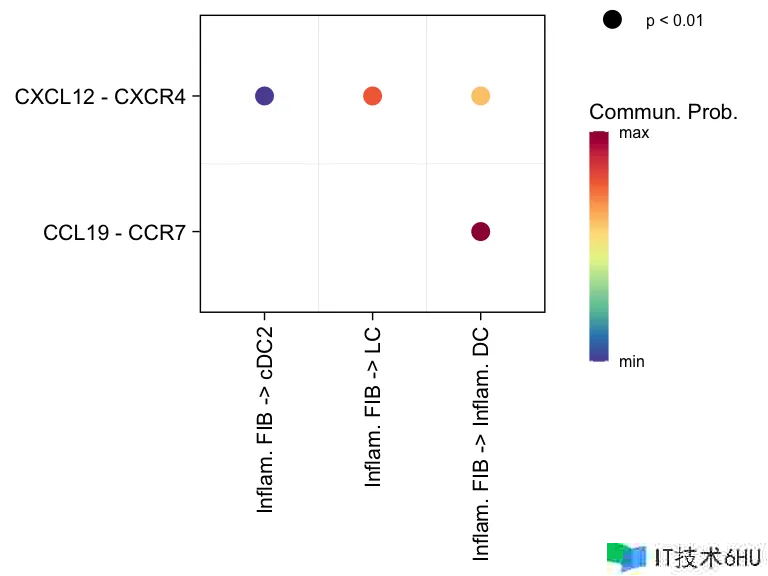
netVisual_bubble(cellchat, sources.use = 4, targets.use = c(5:11), signaling = c("CCL","CXCL"), remove.isolate = FALSE)
#> Comparing communications on a single object
# (3) show all the significant interactions (L-R pairs) based on user's input (defined by `pairLR.use`)
pairLR.use <- extractEnrichedLR(cellchat, signaling = c("CCL","CXCL","FGF"))
netVisual_bubble(cellchat, sources.use = c(3,4), targets.use = c(5:8), pairLR.use = pairLR.use, remove.isolate = TRUE)
#> Comparing communications on a single object
# set the order of interacting cell pairs on x-axis
# (4) Default: first sort cell pairs based on the appearance of sources in levels(object@idents), and then based on the appearance of targets in levels(object@idents)
# (5) sort cell pairs based on the targets.use defined by users
netVisual_bubble(cellchat, targets.use = c("LC","Inflam. DC","cDC2","CD40LG TC"), pairLR.use = pairLR.use, remove.isolate = TRUE, sort.by.target = T)
# (6) sort cell pairs based on the sources.use defined by users
netVisual_bubble(cellchat, sources.use = c("FBN1 FIB","APOE FIB","Inflam. FIB"), pairLR.use = pairLR.use, remove.isolate = TRUE, sort.by.source = T)
# (7) sort cell pairs based on the sources.use and then targets.use defined by users
netVisual_bubble(cellchat, sources.use = c("FBN1 FIB","APOE FIB","Inflam. FIB"), targets.use = c("LC","Inflam. DC","cDC2","CD40LG TC"), pairLR.use = pairLR.use, remove.isolate = TRUE, sort.by.source = T, sort.by.target = T)
# (8) sort cell pairs based on the targets.use and then sources.use defined by users
netVisual_bubble(cellchat, sources.use = c("FBN1 FIB","APOE FIB","Inflam. FIB"), targets.use = c("LC","Inflam. DC","cDC2","CD40LG TC"), pairLR.use = pairLR.use, remove.isolate = TRUE, sort.by.source = T, sort.by.target = T, sort.by.source.priority = FALSE)
(B) Chord diagram
类似于 Bubble plot,CellChat 供给了一个函数 netVisual_chord_gene 来制作 Chord diagram
- 显现从一些细胞群到其他细胞群的一切彼此效果(L-R 对或信号通路)。两种特殊情况:一种显现从一个细胞群发送的一切交互,另一种显现一个细胞群接纳的一切交互。
- 显现用户输入的交互或用户界说的某些信号通路
# show all the significant interactions (L-R pairs) from some cell groups (defined by 'sources.use') to other cell groups (defined by 'targets.use')
# show all the interactions sending from Inflam.FIB
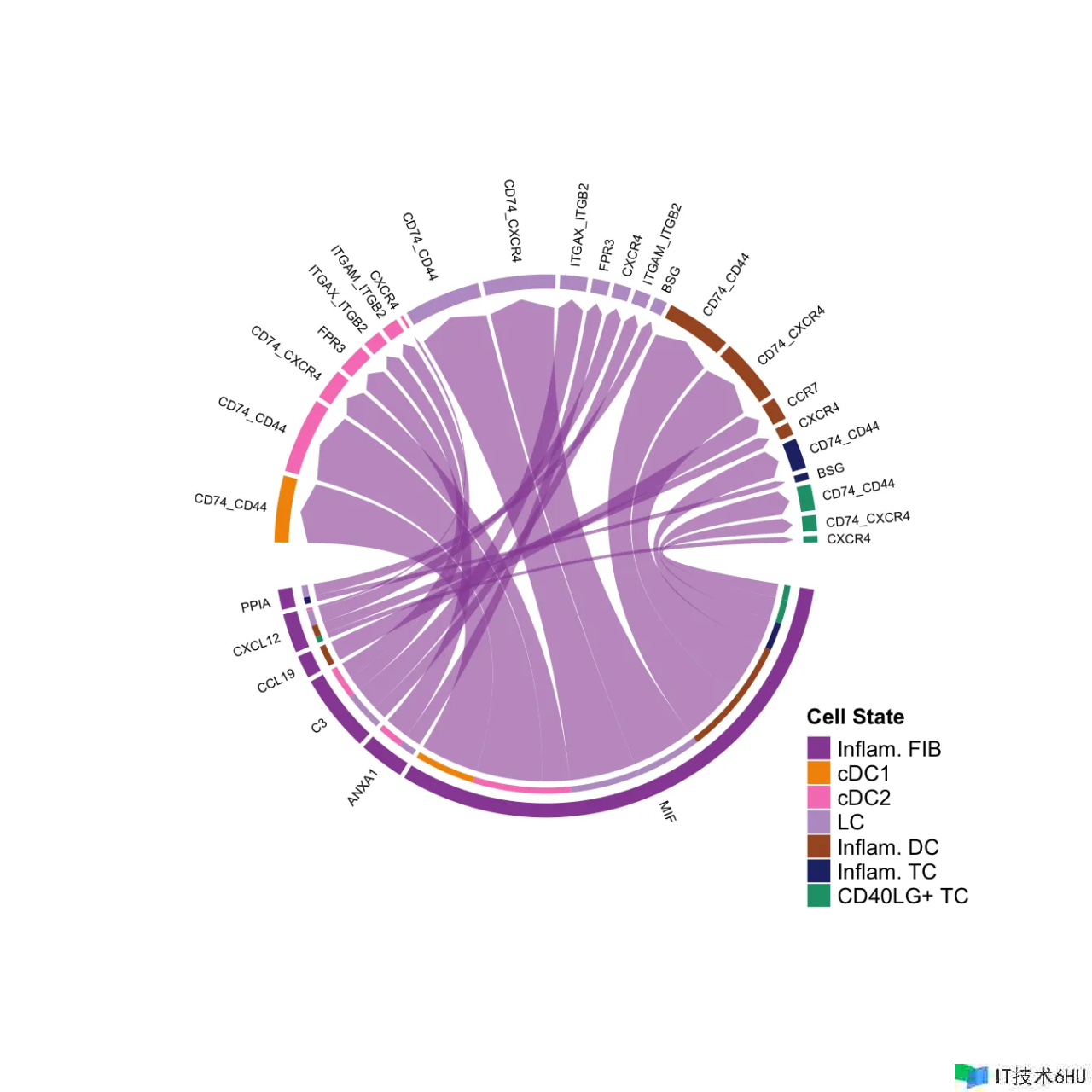
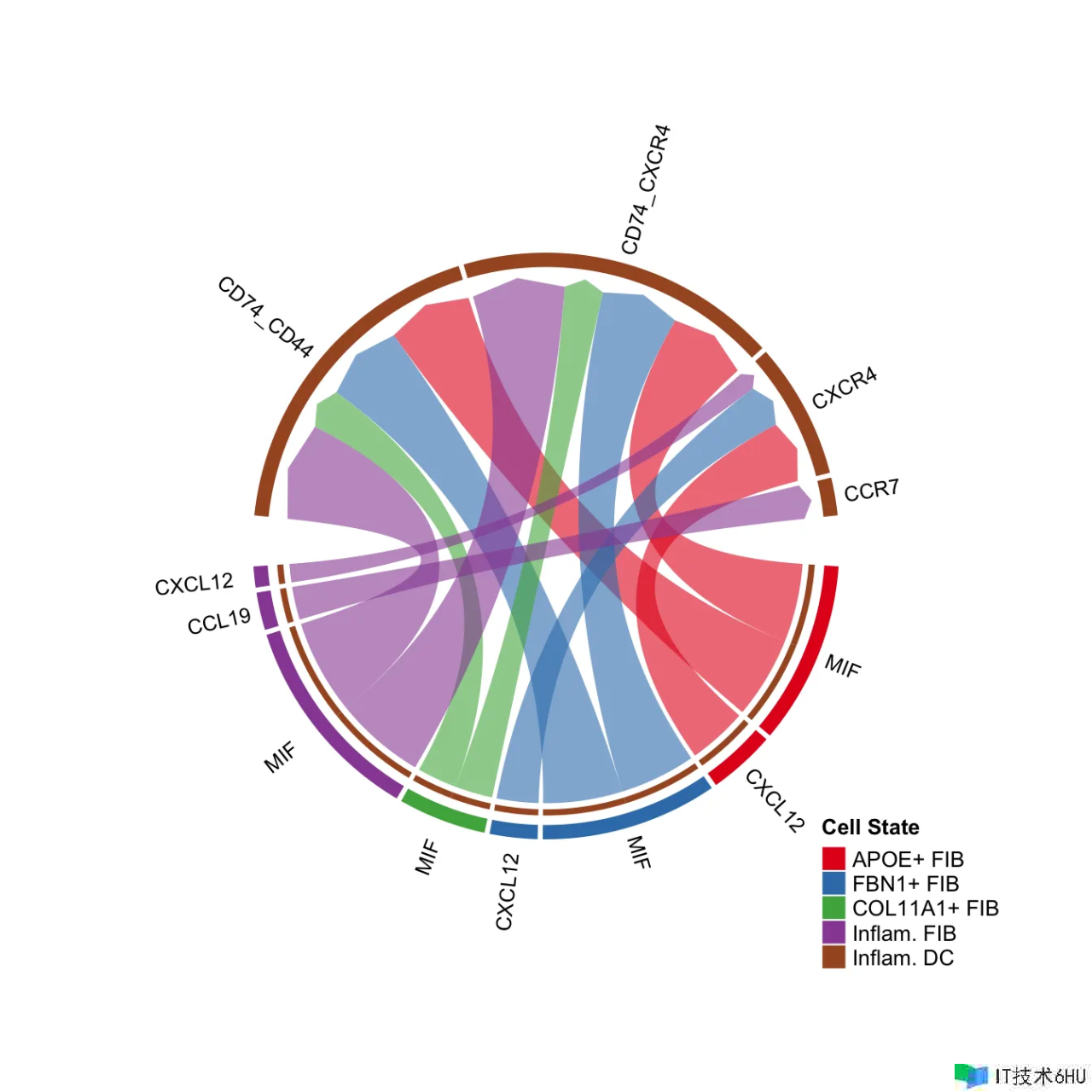
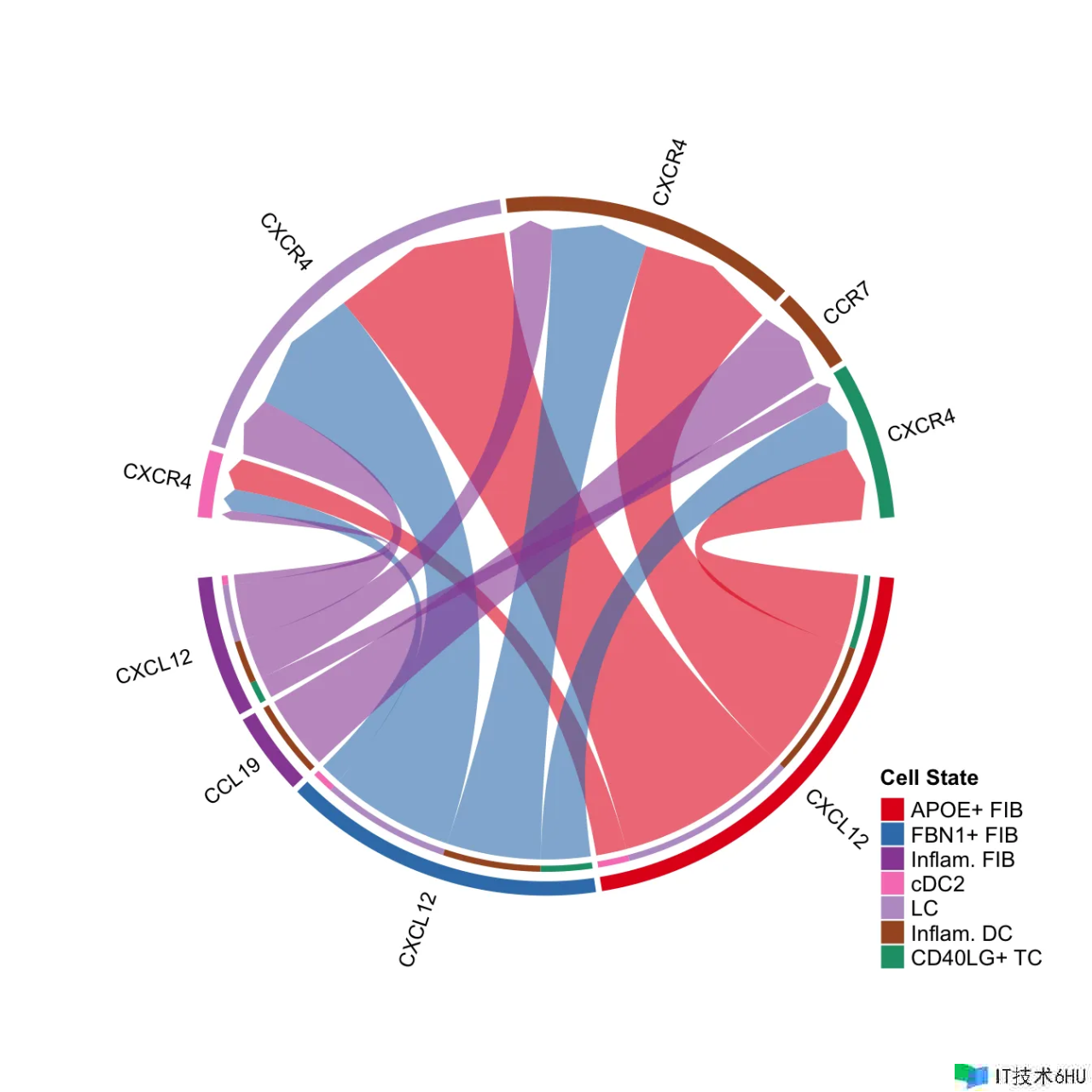
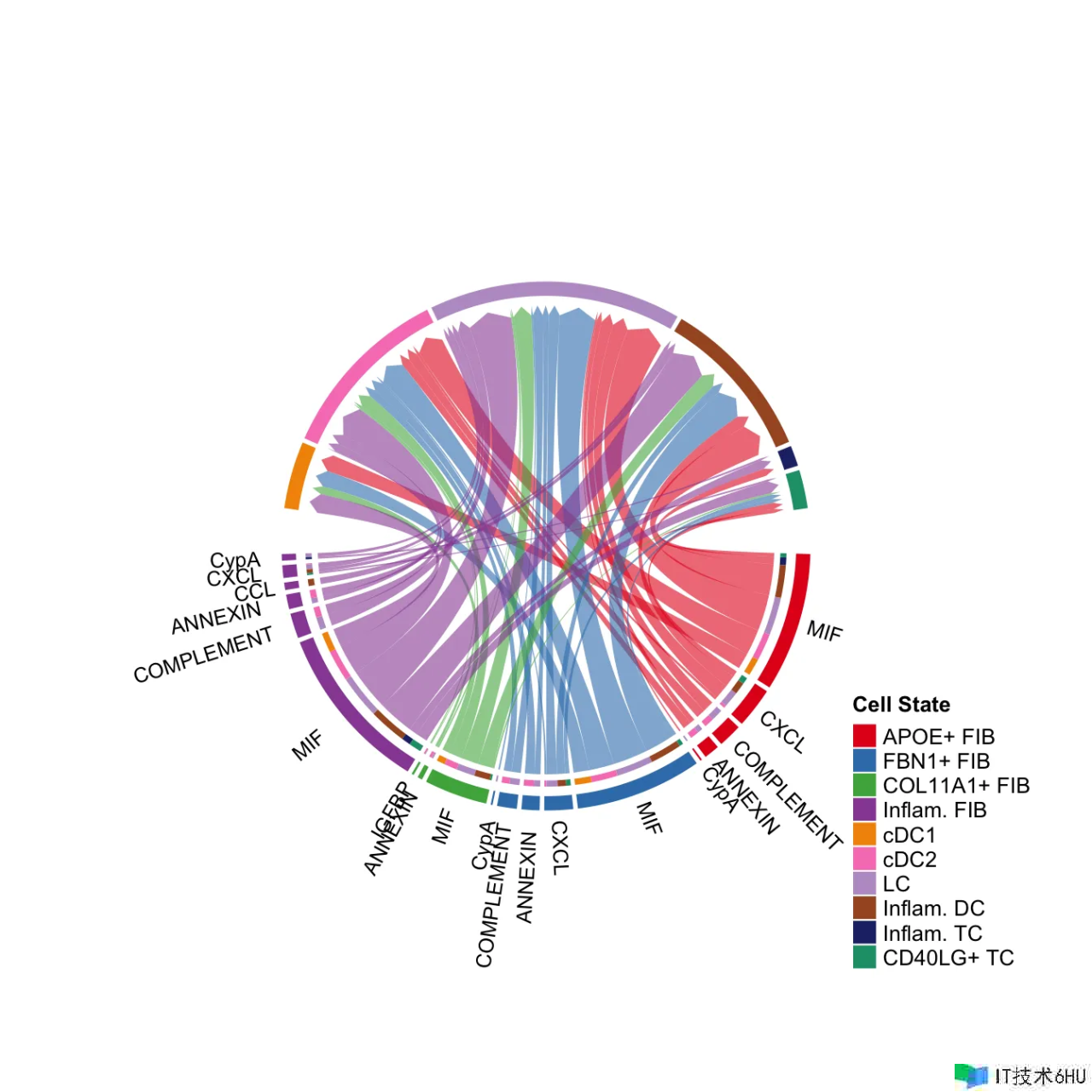
netVisual_chord_gene(cellchat, sources.use = 4, targets.use = c(5:11), lab.cex = 0.5,legend.pos.y = 30)
# show all the interactions received by Inflam.DC
netVisual_chord_gene(cellchat, sources.use = c(1,2,3,4), targets.use = 8, legend.pos.x = 15)
# show all the significant interactions (L-R pairs) associated with certain signaling pathways
netVisual_chord_gene(cellchat, sources.use = c(1,2,3,4), targets.use = c(5:11), signaling = c("CCL","CXCL"),legend.pos.x = 8)
# show all the significant signaling pathways from some cell groups (defined by 'sources.use') to other cell groups (defined by 'targets.use')
netVisual_chord_gene(cellchat, sources.use = c(1,2,3,4), targets.use = c(5:11), slot.name = "netP", legend.pos.x = 10)
留意:在生成绘图时请疏忽注释,例如“留意:第一个链接端是从扇区‘MIF’制作出来的。”。 假如基因名称重叠,您能够经过减小值来调整参数 small.gap。
3.3 运用小提琴/点图制作信号基因表达散布
假如已装置 Seurat R 包,CellChat 能够运用 Seurat 封装函数 plotGeneExpression 制作与配体-受体对或信号通路相关的信号基因的表达散布。该函数供给三种类型的可视化,包括“violin”、“dot”、“bar”。别的,用户还能够运用 extractEnrichedLR 提取与揣度的配体-受体对或信号通路相关的信号基因,然后运用 Seurat 或其他包制作基因表达。
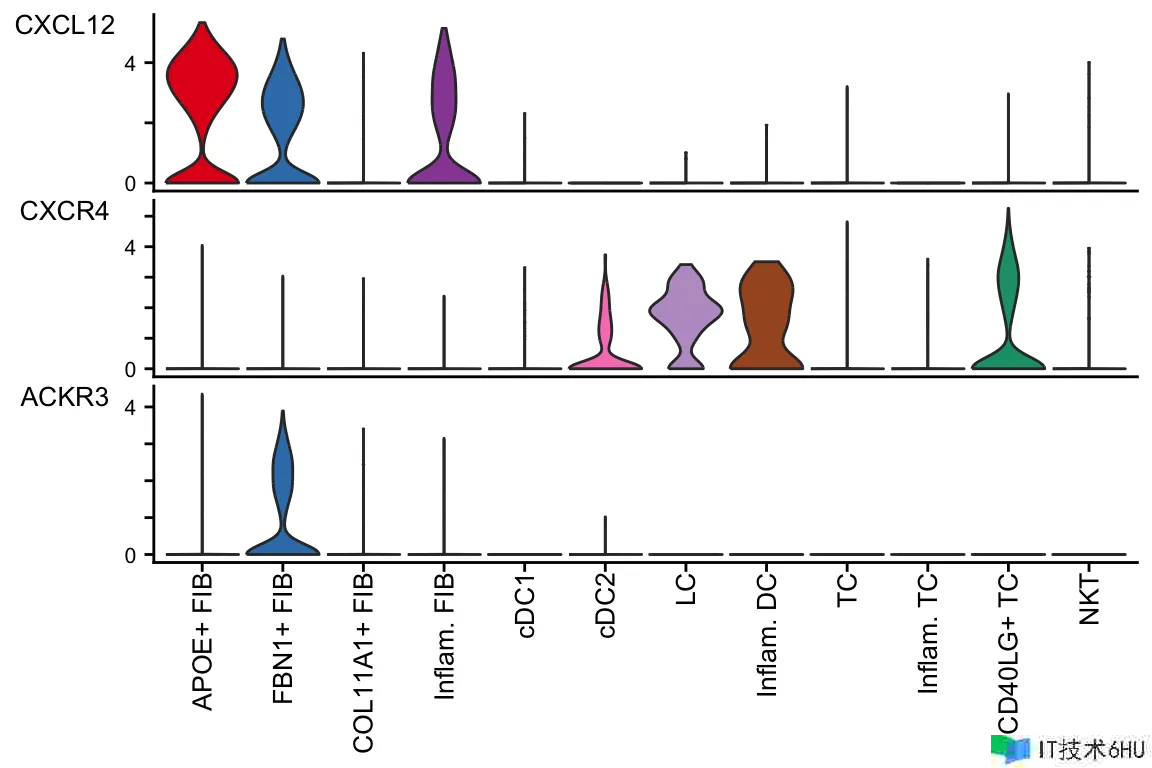
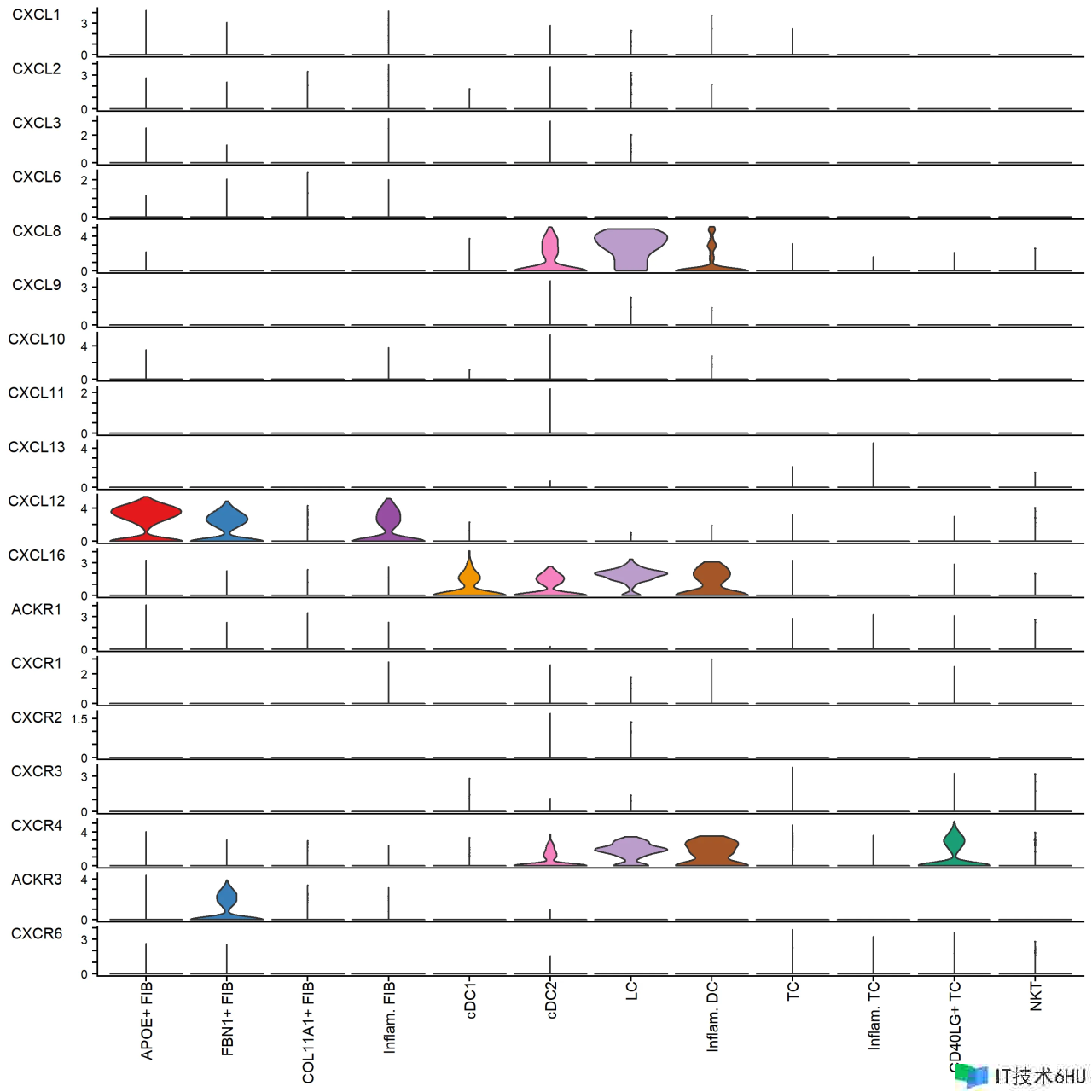
plotGeneExpression(cellchat, signaling = "CXCL", enriched.only = TRUE, type = "violin")
print(as.numeric(execution.time, units = "secs"))
默许情况下,plotGeneExpression 仅显现与揣度的重要通讯相关的信号基因的表达。用户能够经过以下办法显现与一种信号通路相关的一切信号基因的表达
plotGeneExpression(cellchat, signaling = "CXCL", enriched.only = FALSE)
execution.time = Sys.time() - ptm
4 Part IV: 细胞间通讯网络系统剖析
为了便于解说杂乱的细胞间通讯网络,CellChat 经过从图论、形式辨认和流形学习中抽象出来的办法对网络进行定量丈量。
- 它能够运用网络剖析的中心性衡量来确认给定信号网络中的首要信号源(sources)和方针(targets)以及中介(mediators)和影响者(influencers)
- 它能够经过运用形式辨认办法猜测特定细胞类型的要害传入和传出信号以及不同细胞类型之间的和谐呼应。
- 它能够经过界说类似性衡量并从功用和拓扑的角度履行流形学习来对信号通路进行分组。
- 它能够经过多个网络的联合流形学习来描绘保存的和特定于上下文的信号通路。
4.1 确认细胞群的信号效果(例如,首要发送者、接纳者)以及首要的奉献信号
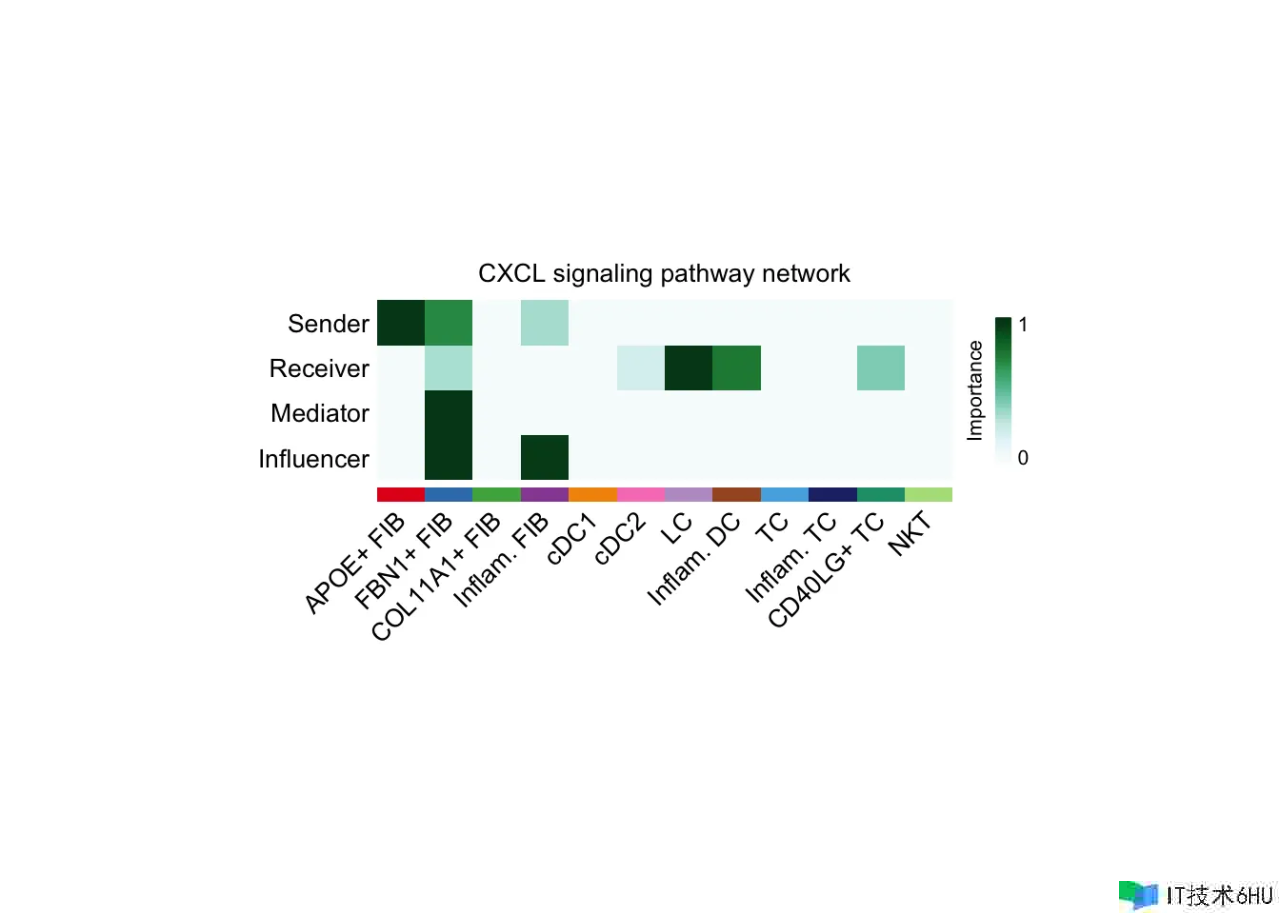
CellChat 经过为每个 cell group 核算多个网络中心性衡量,能够轻松辨认细胞间通讯网络中的首要发送者(senders)、接纳者(receivers)、中介者(mediators)和影响者(influencers)。详细来说,咱们运用加权定向网络中的办法,包括出度(out-degree)、入度(in-degree)、流间性(flow betweenesss)和信息中心性(information centrality),别离辨认细胞间通讯的首要发送者、接纳者、中介者和影响者。在权重为核算的通讯概率的加权有向网络中,出度(outdegree)核算为来自一个细胞组的传出信号的通讯概率之和,入度(in-degree)核算为去到一个细胞组的传入信号的通讯概率之和,可用于别离辨认信号网络的首要细胞发送者和接纳者。关于流介数(flow betweenness)和信息中心性(information centrality)的界说,请查看 CellChat 的论文和相关参阅资料。
用户能够在热图(选项 A)和二维图(选项 B)上可视化中心性分数。CellChat 还能够答复某些细胞组的 outgoing 或 incoming 信号中哪些信号奉献最大的问题(选项 C)。
(A) 核算和可视化网络中心性分数
ptm = Sys.time()
# Compute the network centrality scores
cellchat <- netAnalysis_computeCentrality(cellchat, slot.name = "netP") # the slot 'netP' means the inferred intercellular communication network of signaling pathways
# Visualize the computed centrality scores using heatmap, allowing ready identification of major signaling roles of cell groups
netAnalysis_signalingRole_network(cellchat, signaling = pathways.show, width = 8, height = 2.5, font.size = 10)
(B) 可视化 2D 空间中的首要发送者(sources)和接纳者(targets)
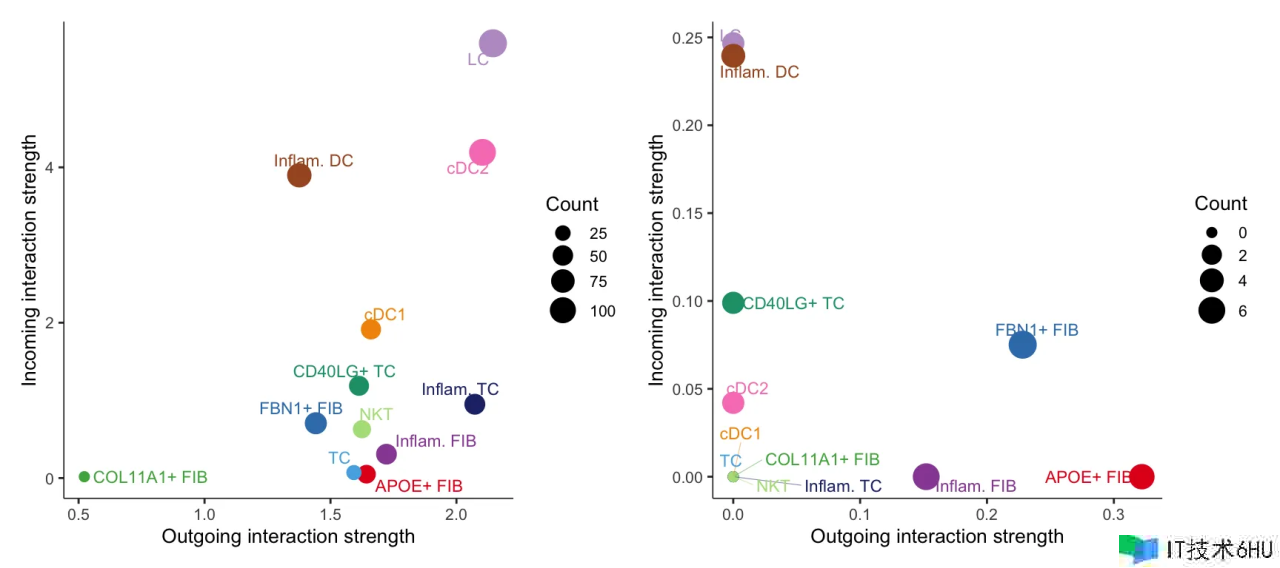
CellChat 还供给了另一种直观的办法来运用散点图在二维空间中可视化首要的发送者(sources)和接纳者(targets)。x 轴和 y 轴别离是与每个细胞组相相关的总 outgoing 或 incoming 通讯概率。点的巨细与与每个细胞组相相关的揣度链接数(outgoing and incoming)成份额。点的颜色标明不同的 cell groups。假如界说了group,则点的形状标明不同类别的细胞组。
# Signaling role analysis on the aggregated cell-cell communication network from all signaling pathways
gg1 <- netAnalysis_signalingRole_scatter(cellchat)
#> Signaling role analysis on the aggregated cell-cell communication network from all signaling pathways
# Signaling role analysis on the cell-cell communication networks of interest
gg2 <- netAnalysis_signalingRole_scatter(cellchat, signaling = c("CXCL", "CCL"))
#> Signaling role analysis on the cell-cell communication network from user's input
gg1 gg2
(C) 确认对某些 cell groups 的 outgoing 或 incoming 信号奉献最大的信号
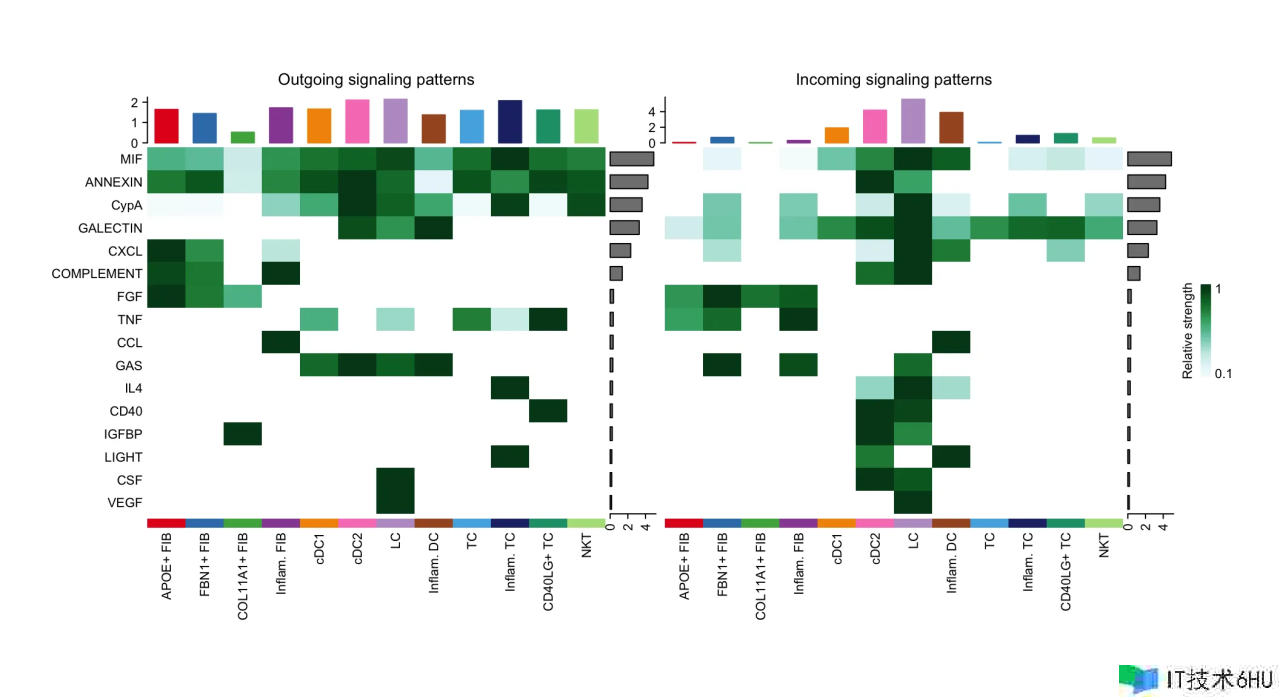
咱们还能够答复哪些信号对某些 cell groups 的 outgoing 或 incoming 信号奉献最大的问题。在这个热图中,颜色条代表一个信号通路在 cell groups 间的相对信号强度(注:值经过 row-scaled)。顶部有色条形图显现了经过总结热图中显现的一切信号通路来标明 cell group 的总信号强度。右侧的灰色条形图显现了经过总结热图中显现的一切 cell group 来标明信号通路的总信号强度。
# Signaling role analysis on the aggregated cell-cell communication network from all signaling pathways
ht1 <- netAnalysis_signalingRole_heatmap(cellchat, pattern = "outgoing")
ht2 <- netAnalysis_signalingRole_heatmap(cellchat, pattern = "incoming")
ht1 ht2
# Signaling role analysis on the cell-cell communication networks of interest
ht <- netAnalysis_signalingRole_heatmap(cellchat, signaling = c("CXCL", "CCL"))
4.2 辨认全局通讯形式以探究多种细胞类型和信号通路怎么和谐在一起
除了探究各个通路的详细通讯之外,一个重要的问题是多个 cell groups 和信号通路怎么和谐发挥效果。CellChat 采用形式辨认办法来辨认全局通讯形式。
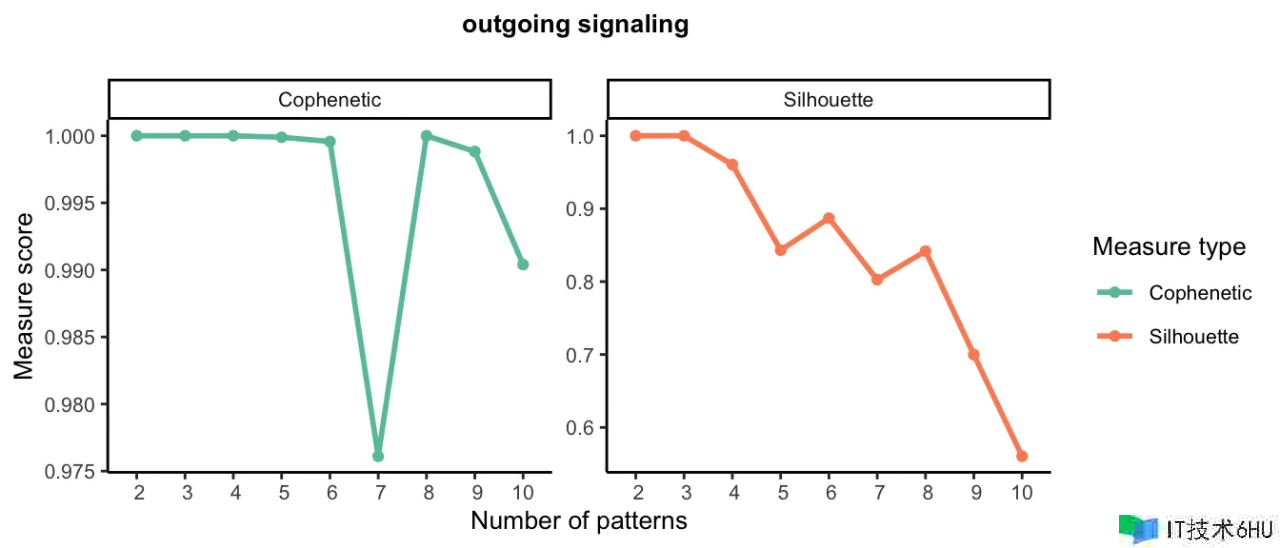
随着形式数量的增加,或许会呈现冗余形式,然后难以解说通讯形式。CellChat 默许选择了五种形式。一般来说,形式的数量大于 2 才具有生物学含义。此外,CellChat 还供给了一个函数 selectK 来揣度形式的数量,它依据 NMF R 包中现已完成的两个方针,包括 Cophenetic 和 Silhouette。这两个方针都依据共识矩阵的层次聚类来衡量特定数量形式的稳定性。关于必定数量的形式,合适数量的形式是 Cophenetic 和 Silhouette 值开始忽然下降的形式。
这项剖析能够针对 outgoing(选项 A)和 incoming(选项 B)信号形式进行。Outgoing 形式显现了发送细胞(即 cells as signal source)怎么彼此和谐以及它们怎么与特定信号通路和谐以推进通讯。Incoming 形式显现了方针细胞(即 cells as signal receivers)怎么彼此和谐以及它们怎么与特定信号通路和谐以呼应入向信号。
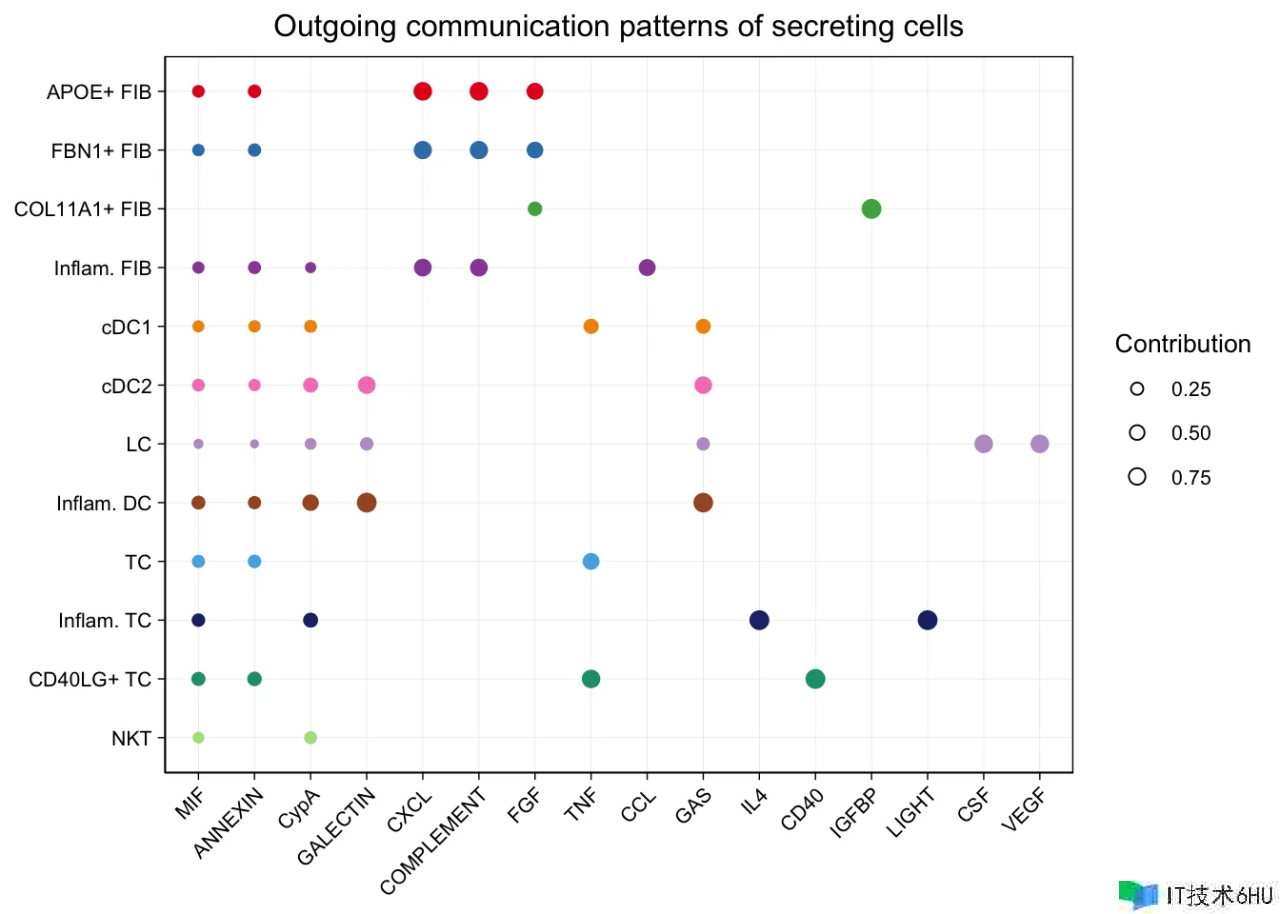
(A) 确认并可视化排泄细胞的 outgoing 通讯形式
Outgoing patterns 提醒了 sender cells(i.e. cells as signal source)怎么彼此和谐以及它们怎么与某些信号通路和谐以驱动通讯。
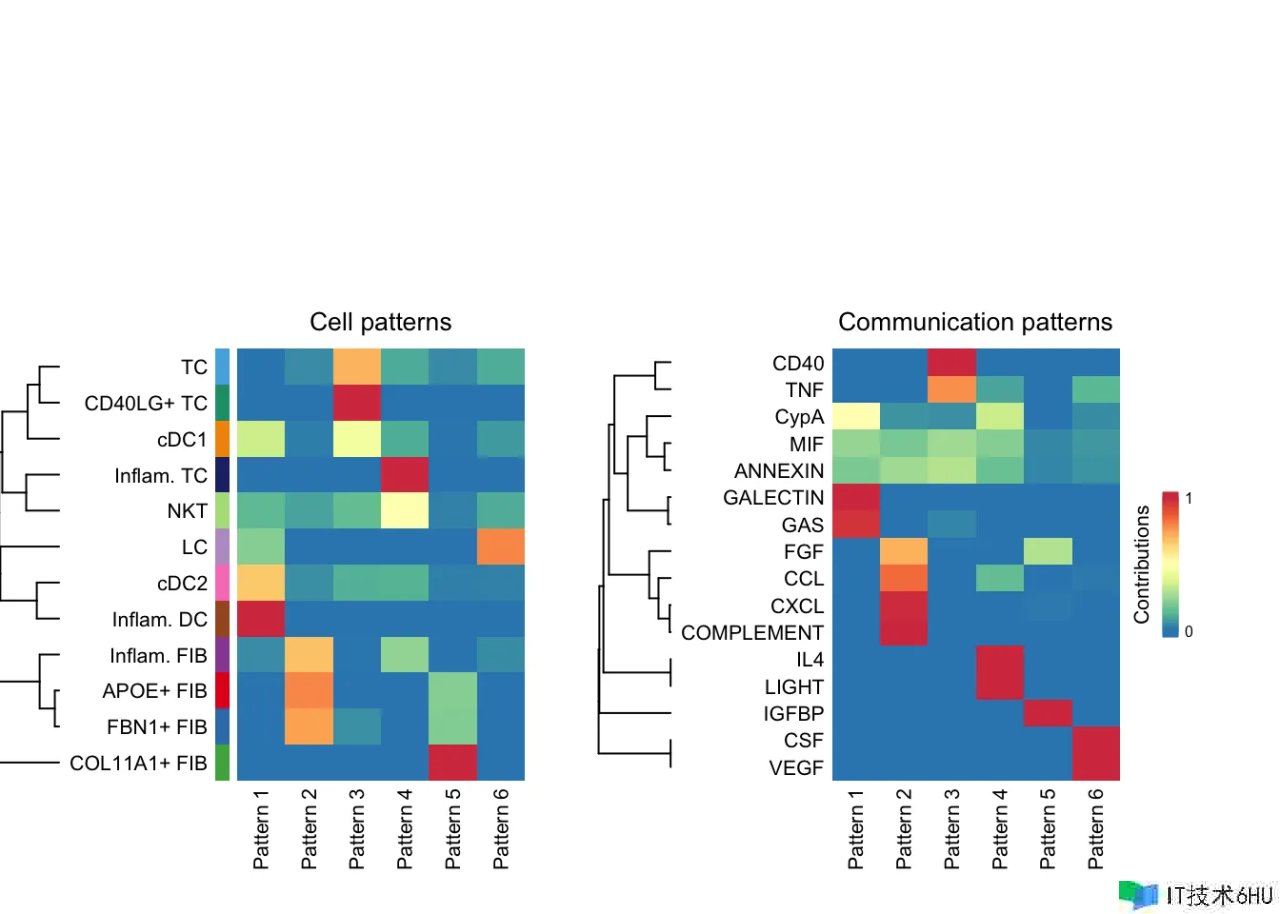
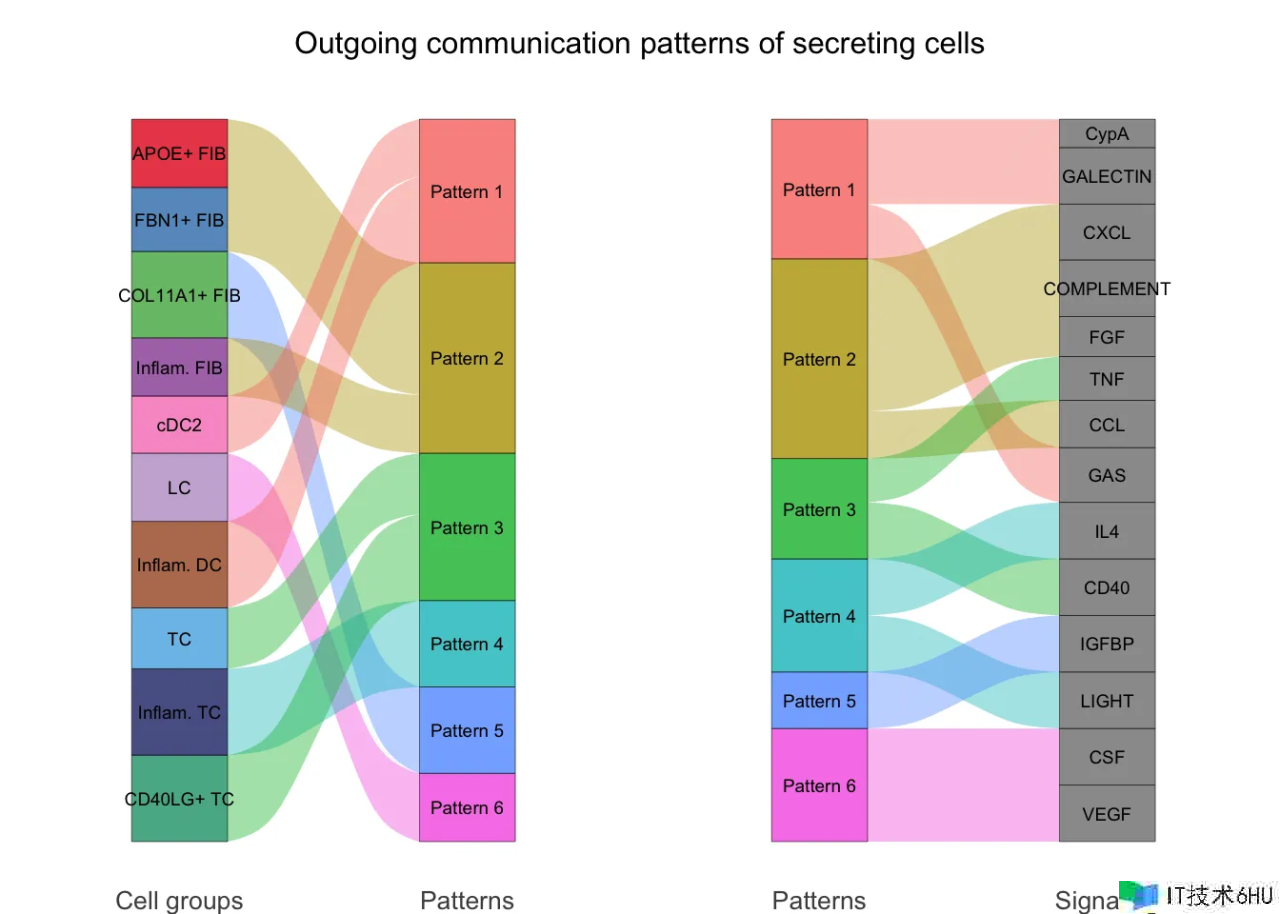
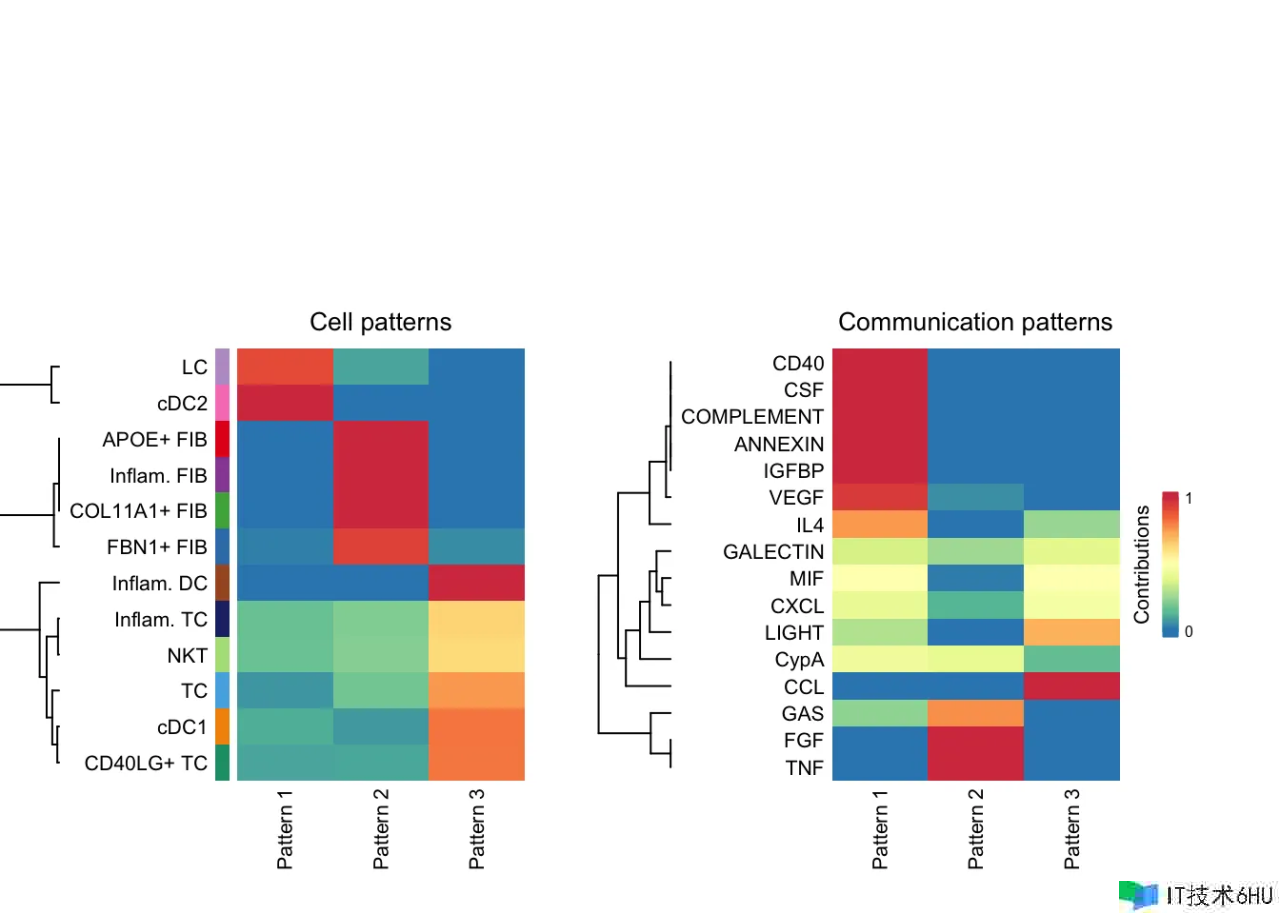
关于 outgoing (or incoming) patterns,从外向(或入向)细胞间通讯概率的矩阵分解中输出的细胞组形式矩阵 W 标明这些细胞组怎么和谐发送(或接纳)信号,信号通路形式矩阵 H 标明这些信号通路怎么和谐作业以发送(或接纳)信号。为了直观显现潜在形式与细胞组和配体-受体对或信号通路的相关,咱们运用了一种河流(alluvial)图。咱们首先将 W 的每一行和 H 的每一列归一化为 [0,1],然后假如它们小于阈值(默许值为 0.5),则将 W 和 H 中的元素设为零。这种阈值处理答应提醒与每个揣度形式相关的最丰厚的细胞组和信号通路。这些经过阈值处理的矩阵 W 和 H 被用作创立河流图的输入。
为了直接将细胞群与其丰厚的信号通路联络起来,咱们将 W 和 H 中的元素设置为零,假如它们小于 1/R,其间 R 是潜在形式的数量。经过运用不太严厉的阈值,能够获得与每个细胞群相关的更丰厚的信号通路。 运用经过将 W 乘以 H 核算得出的每个细胞群对每个信号通路的奉献分数,咱们构建了一个点图,其间点巨细与奉献分数成份额,以显现细胞群与其丰厚的信号通路之间的相关。用户还能够下降参数截止值,以显现与每个细胞群相关的更丰厚的信号通路。
加载通讯形式剖析所需的包
library(NMF)
library(ggalluvial)
在这儿,咱们运转 selectK 来揣度形式的数量。
selectK(cellchat, pattern = "outgoing")
当传出形式的数量为 6 时,Cophenetic 和 Silhouette 值都开始忽然下降。
nPatterns = 6
cellchat <- identifyCommunicationPatterns(cellchat, pattern = "outgoing", k = nPatterns)
# river plot
netAnalysis_river(cellchat, pattern = "outgoing")
#> Please make sure you have load `library(ggalluvial)` when running this function
# dot plot
netAnalysis_dot(cellchat, pattern = "outgoing")
(B) 辨认并可视化方针细胞的传入通讯形式
传入形式显现了方针细胞(i.e. cells as signal receivers)怎么彼此和谐以及它们怎么与某些信号通路和谐以呼应传入信号。
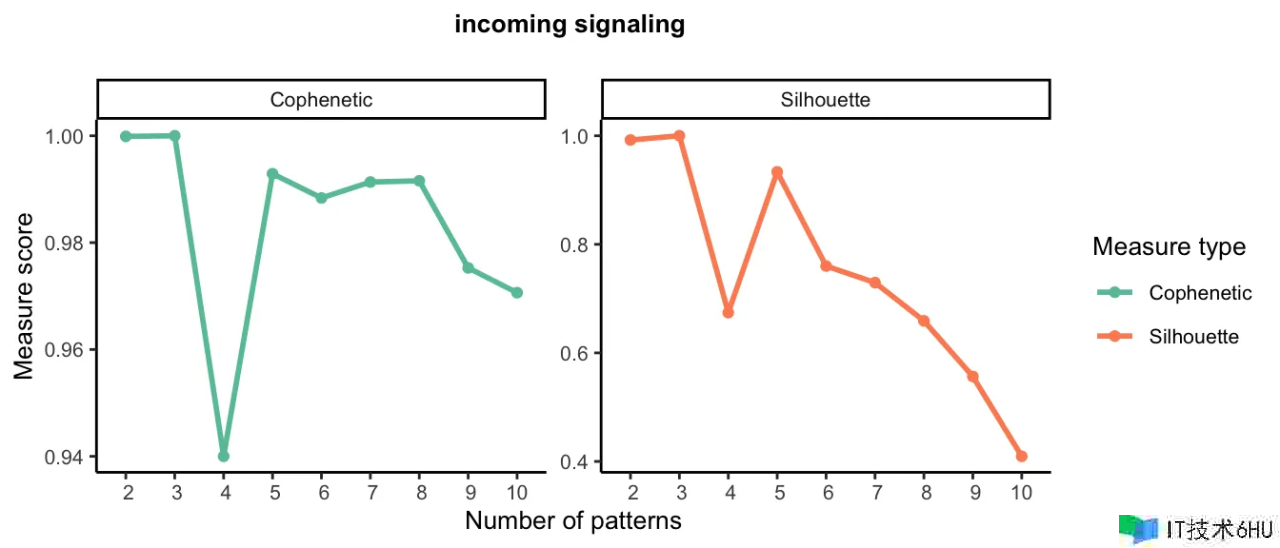
selectK(cellchat, pattern = "incoming")
当传入形式的数量为 3 时,Cophenetic 值开始下降。
nPatterns = 3
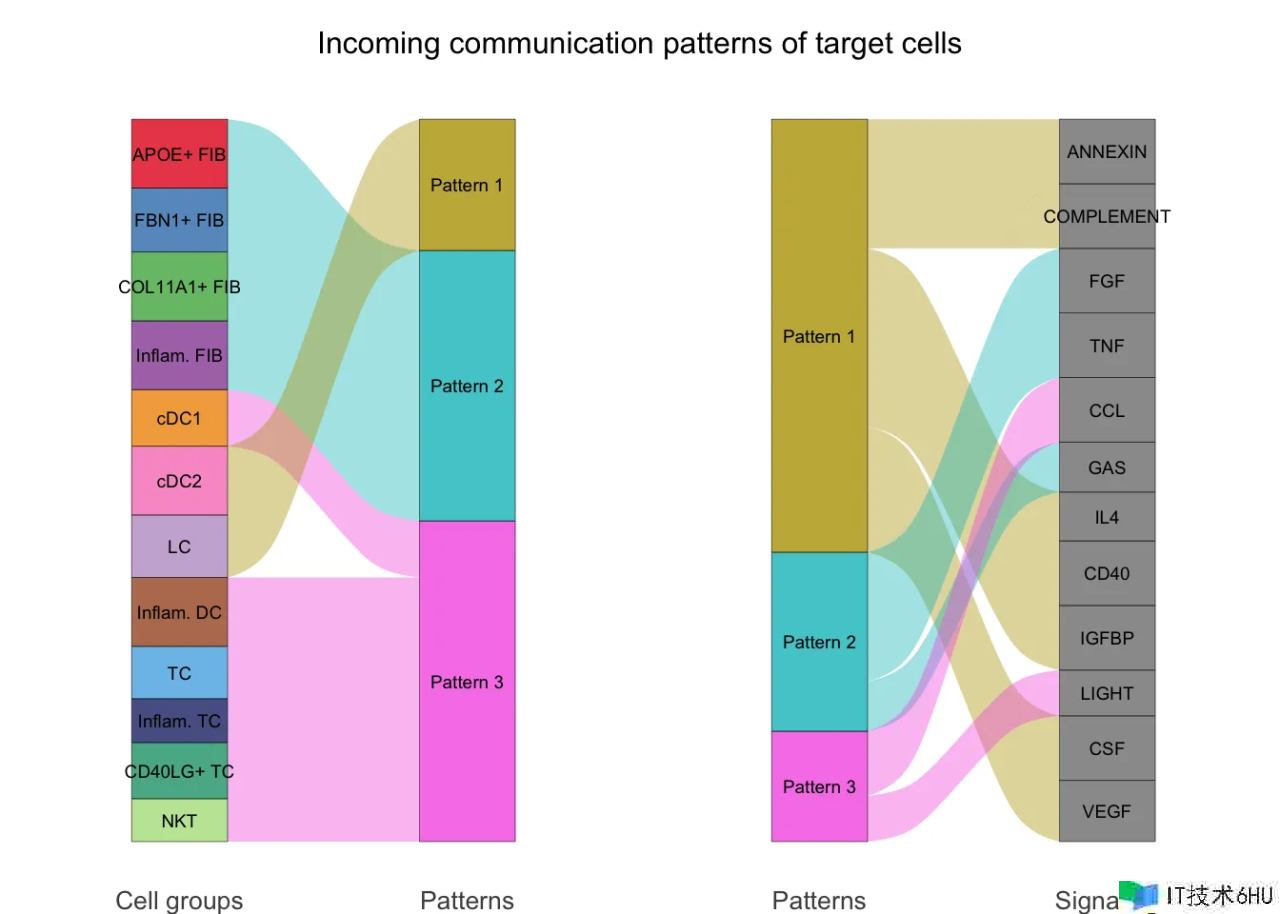
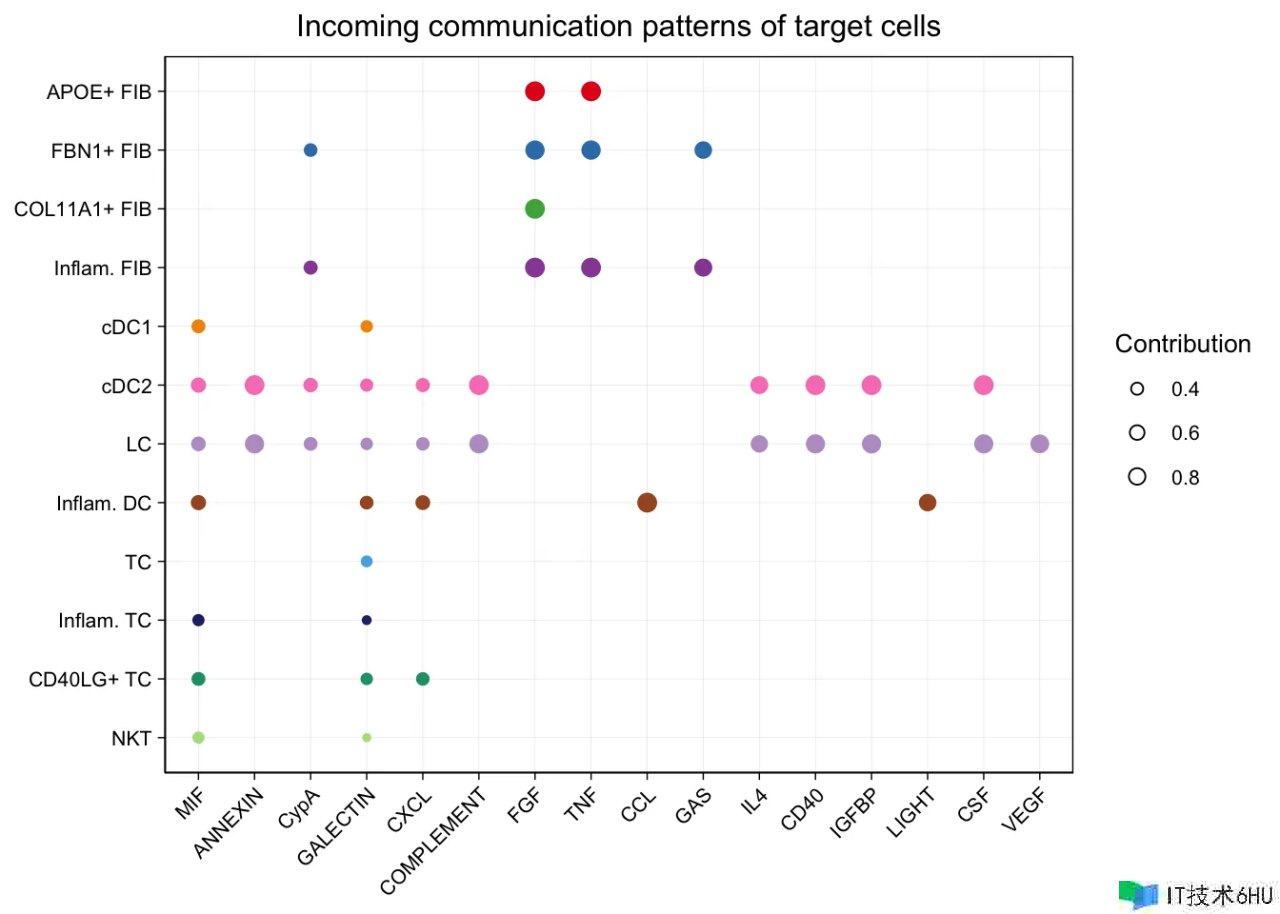
cellchat <- identifyCommunicationPatterns(cellchat, pattern = "incoming", k = nPatterns)
# river plot
netAnalysis_river(cellchat, pattern = "incoming")
#> Please make sure you have load `library(ggalluvial)` when running this function
# dot plot
netAnalysis_dot(cellchat, pattern = "incoming")
4.3 信号网络的流形和分类学习剖析
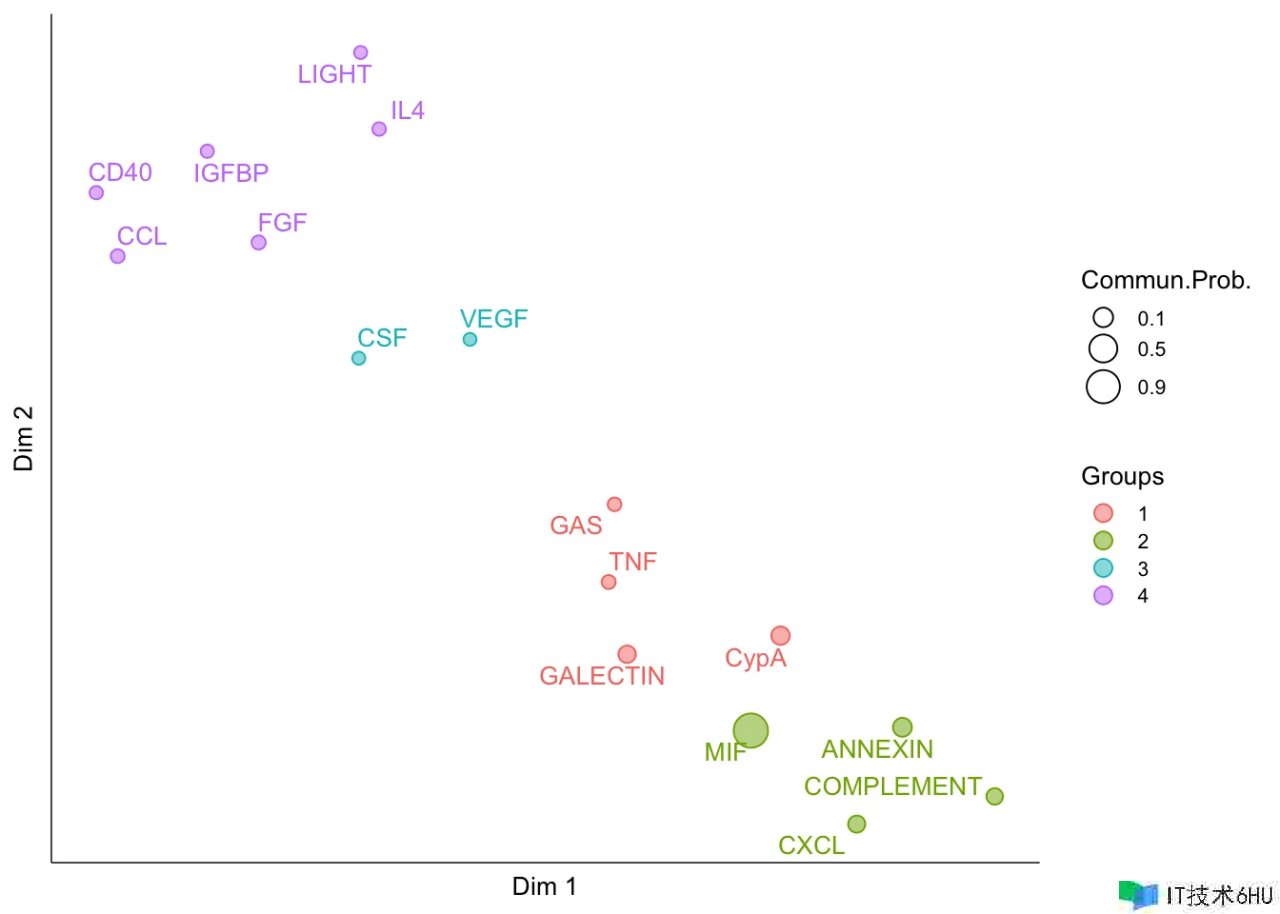
此外,CellChat 能够量化一切重要信号通路之间的类似性,然后依据它们的细胞通讯网络类似性对它们进行分组。能够依据功用(Functional)或结构(Structural)类似性进行分组。
Functional similarity:高度的功用类似性标明首要的发送者(senders)和接纳者(receivers)类似,能够解说为两个信号通路或两个配体-受体对体现出类似和/或冗余的效果。功用类似性剖析需求两个数据集之间具有相同的细胞群组成。
Structural similarity:结构类似性用来比较它们的信号网络结构,不考虑发送者和接纳者的类似性。
依据功用类似性辨认信号基团
cellchat <- computeNetSimilarity(cellchat, type = "functional")
cellchat <- netEmbedding(cellchat, type = "functional")
#> Manifold learning of the signaling networks for a single dataset
cellchat <- netClustering(cellchat, type = "functional")
#> Classification learning of the signaling networks for a single dataset
# Visualization in 2D-space
netVisual_embedding(cellchat, type = "functional", label.size = 3.5)
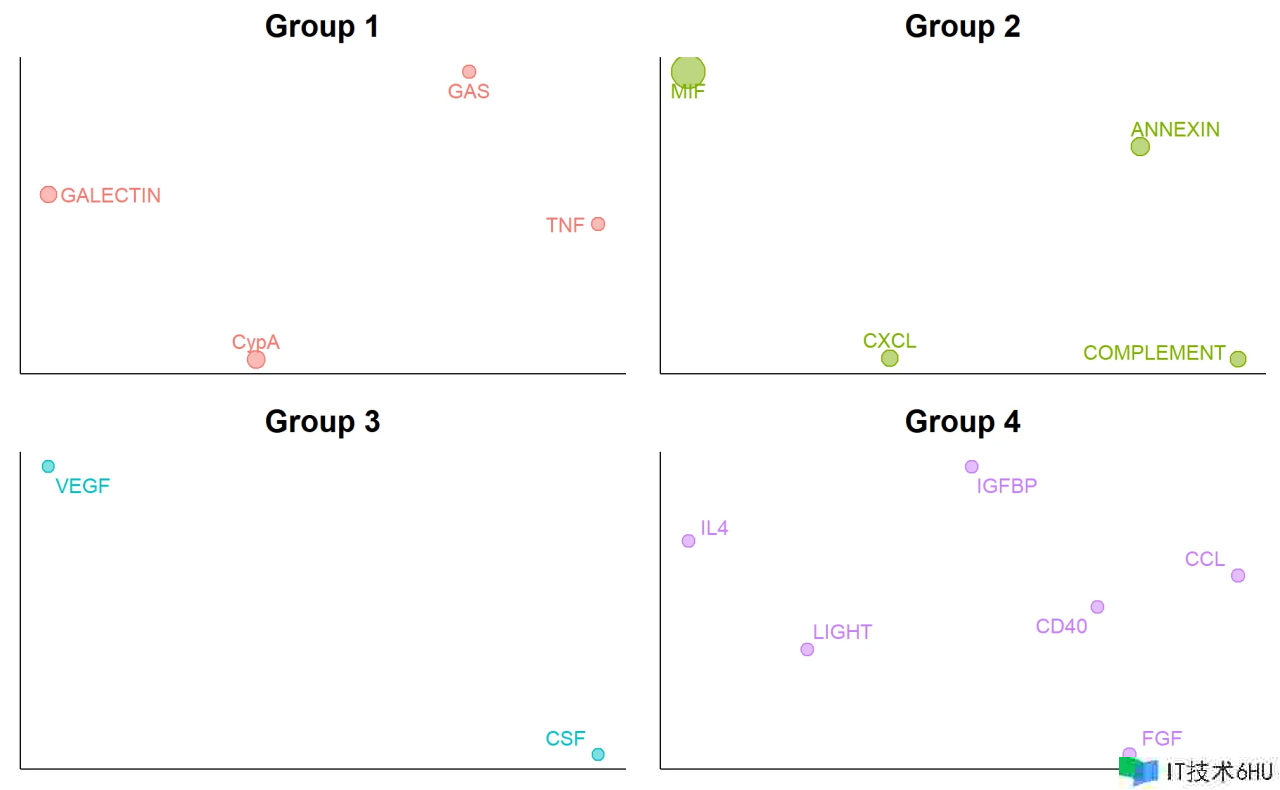
netVisual_embeddingZoomIn(cellchat, type = "functional", nCol = 2)
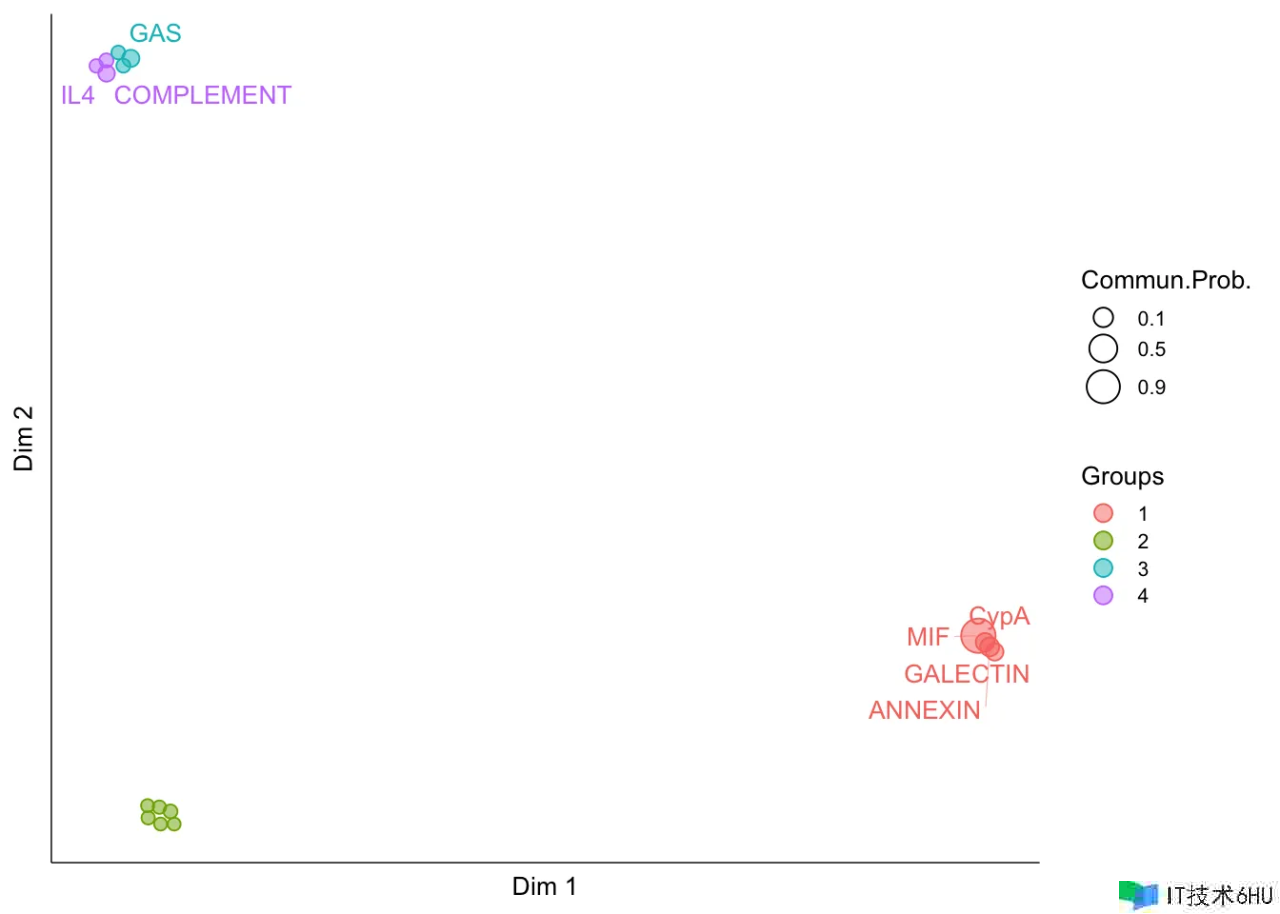
依据结构类似性辨认信号基团
cellchat <- computeNetSimilarity(cellchat, type = "structural")
cellchat <- netEmbedding(cellchat, type = "structural")
#> Manifold learning of the signaling networks for a single dataset
cellchat <- netClustering(cellchat, type = "structural")
#> Classification learning of the signaling networks for a single dataset
# Visualization in 2D-space
netVisual_embedding(cellchat, type = "structural", label.size = 3.5)
netVisual_embeddingZoomIn(cellchat, type = "structural", nCol = 2)
execution.time = Sys.time() - ptm
print(as.numeric(execution.time, units = "secs"))
5 Part V: 保存 CellChat 方针
saveRDS(cellchat, file = "cellchat_humanSkin_LS.rds")
6 Part VI: 经过交互式 CellChat Explorer 探究细胞间通讯
关于单细胞转录组学的 CellChat 剖析,请保证 object@dr 包括数据的低维空间,例如“umap”和“tsne”,以便生成信号基因的特征图。能够经过函数 addReduction 增加新的低维空间。
runCellChatApp(cellchat)
完毕
注:本文为个人学习笔记,仅供大家参阅学习,不得用于任何商业意图。如有侵权,请联络作者删去。
本文由mdnice多渠道发布